Acelerando a descoberta de medicamentos: de arquivos FASTA a percepções de GenAI no Databricks
Como criar um pipeline de ponta a ponta que combina Engenharia de Dados, Modelos de Linguagem de Proteínas e GenAI na Databricks Platform.
Publicado: 4 de fevereiro de 2026
por Ram Goli e May Merkle-Tan
Summary
- Processar dados biológicos em escala usando Lakeflow Declarative Pipelines para transformar sequências brutas de proteínas FASTA em tabelas prontas para análise no Unity Catalog.
- Classificar proteínas com modelos transformer aproveitando o ProtBERT, um modelo de linguagem de proteína, para identificar proteínas de transporte de membrana — principais alvos de medicamentos.
- Consulte query de percepções de proteínas em linguagem natural por meio de AI Functions que conectam LLMs diretamente aos seus dados, permitindo que os pesquisadores explorem candidatos a medicamentos promissores de forma conversacional.
O desenvolvimento de fármacos é notoriamente lento e caro. O ciclo de vida médio de Pesquisa e Desenvolvimento (P&D) abrange de 10 a 15 anos, com uma parte significativa dos candidatos falhando durante os ensaios clínicos. Um grande gargalo tem sido a identificação das proteínas-alvo certas no início do processo.
As proteínas são as "moléculas de trabalho" dos organismos vivos — elas catalisam reações, transportam moléculas e atuam como alvos para a maioria dos fármacos modernos. A capacidade de classificar rapidamente as proteínas, entender suas propriedades e identificar candidatos pouco pesquisados poderia acelerar drasticamente o processo de descoberta (por exemplo, Wozniak et al., 2024, Nature Chemical Biology).
É aqui que a convergência da engenharia de dados, do machine learning (ML) e da IA generativa se torna transformadora. Na verdade, você pode criar todo este pipeline em uma única plataforma: a Databricks Data Intelligence Platform.
O que estamos construindo
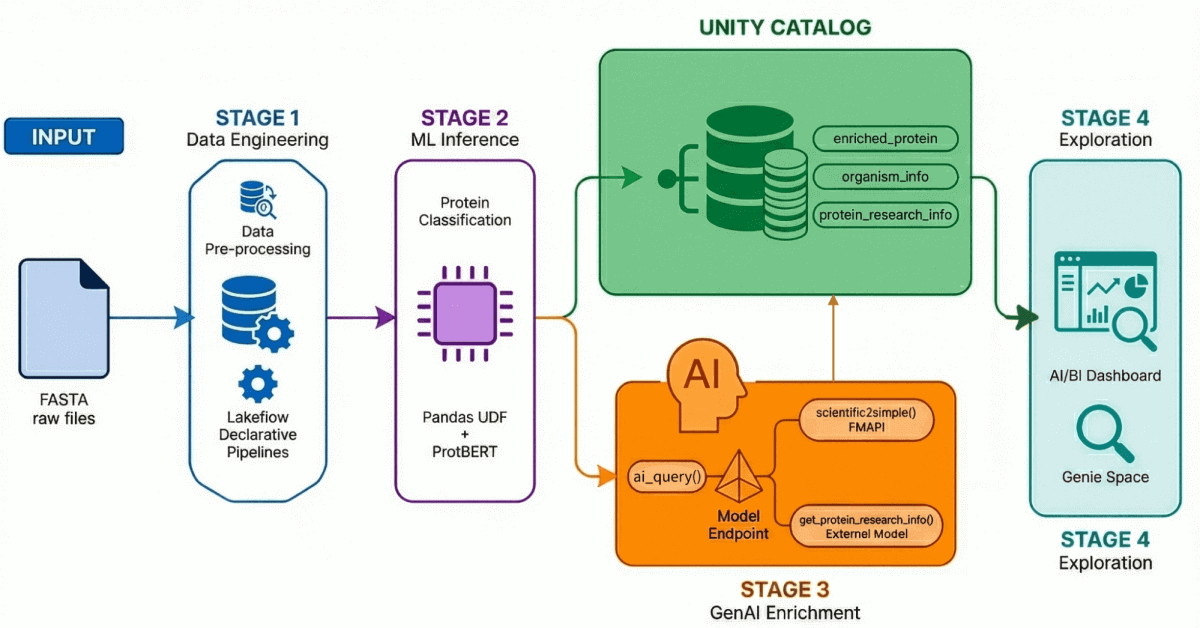
Nosso Acelerador de Soluções para Descoberta de Medicamentos Orientada por IA demonstra um fluxo de trabalho de ponta a ponta por meio de quatro processos principais:
- Ingestão de dados e processamento: mais de 500.000 sequências de proteínas são ingeridas e processadas do UniProt.
- Classificação por IA: um modelo transformer é usado para classificar essas proteínas como hidrossolúveis ou de transporte de membrana.
- Geração de percepções: os dados de proteínas são enriquecidos com percepções de pesquisa geradas por LLM.
- Exploração em Linguagem Natural: Todos os dados processados e enriquecidos são disponibilizados por meio de um painel e ambiente habilitados para IA que suportam consultas em linguagem natural.
Vamos percorrer cada etapa:
Etapa 1: Engenharia de dados com Lakeflow Declarative Pipelines
Os dados biológicos brutos raramente chegam em um formato limpo e pronto para análise. Nossos dados de origem vêm como arquivos FASTA, um formato padrão para representar sequências de proteínas que se parece com isto:
Para um olho não treinado, esses dados de sequência são quase impossíveis de interpretar: uma densa cadeia de códigos de aminoácidos de letra única. No entanto, ao final deste pipeline, os pesquisadores podem consultar esses mesmos dados em linguagem natural, fazendo perguntas como "Mostre-me proteínas de membrana pouco pesquisadas em humanos com alta confiança de classificação" e recebendo percepções acionáveis em troca.
Usando o Lakeflow Declarative Pipelines, criamos uma arquitetura medallion que refina progressivamente esses dados:
- Camada Bronze: Ingestão bruta de arquivos FASTA usando BioPython, extraindo IDs e sequências.
- Camada Prata: análise e estruturação — extraímos nomes de proteínas, informações de organismos, nomes de genes e outros metadados usando transformações regex.
- Camada Gold/Enriquecida: Dados curados e prontos para análise, enriquecidos com métricas derivadas, como peso molecular — prontos para dashboards, modelos de ML e pesquisas posteriores. Esta é a camada confiável que analistas e cientistas query diretamente.
O resultado: Dados de proteínas limpos e governados no Unity Catalog, prontos para ML e analítica posteriores. Fundamentalmente, a linhagem de dados que se estende além desta etapa para as outras (destacadas abaixo) agrega um valor incrível para a reprodutibilidade científica.
Estágio 2: classificação de proteínas com modelos Transformer
Nem todas as proteínas são iguais quando se trata da descoberta de medicamentos. Proteínas de transporte de membrana — aquelas incorporadas nas membranas celulares — são alvos de medicamentos particularmente importantes porque controlam o que entra e sai das células.
Utilizamos o ProtBERT-BFD, um modelo de linguagem de proteína baseado em BERT do Rostlab, com ajuste fino específico para a classificação de proteínas de membrana. Este modelo trata as sequências de aminoácidos como linguagem, aprendendo relações contextuais entre resíduos para prever a função da proteína.
O modelo gera uma classificação (como Membrana ou Solúvel) juntamente com uma pontuação de confiança, que gravamos de volta no Unity Catalog para filtragem e análise posteriores.
Etapa 3: Enriquecimento de dados com GenAI
A classificação nos diz o que é uma proteína. Mas os pesquisadores precisam saber por que isso importa: qual é a pesquisa recente? Onde estão as lacunas? Este é um alvo de medicamento pouco explorado?
É aqui que entram os LLMs. Utilizando tanto a API do Foundational Model do Databricks quanto os endpoints de Modelo Externo, criamos Funções de IA registradas que enriquecem os registros de proteína com contexto de pesquisa.
Etapa 4: Exploração de linguagem natural
Reunimos tudo em um dashboard de IA/BI com o Genie Space ativado.
Os pesquisadores agora podem:
- Filtrar proteínas por organismo, pontuação de classificação e tipo de proteína
- Explorar distribuições de pesos moleculares e confiança de classificação
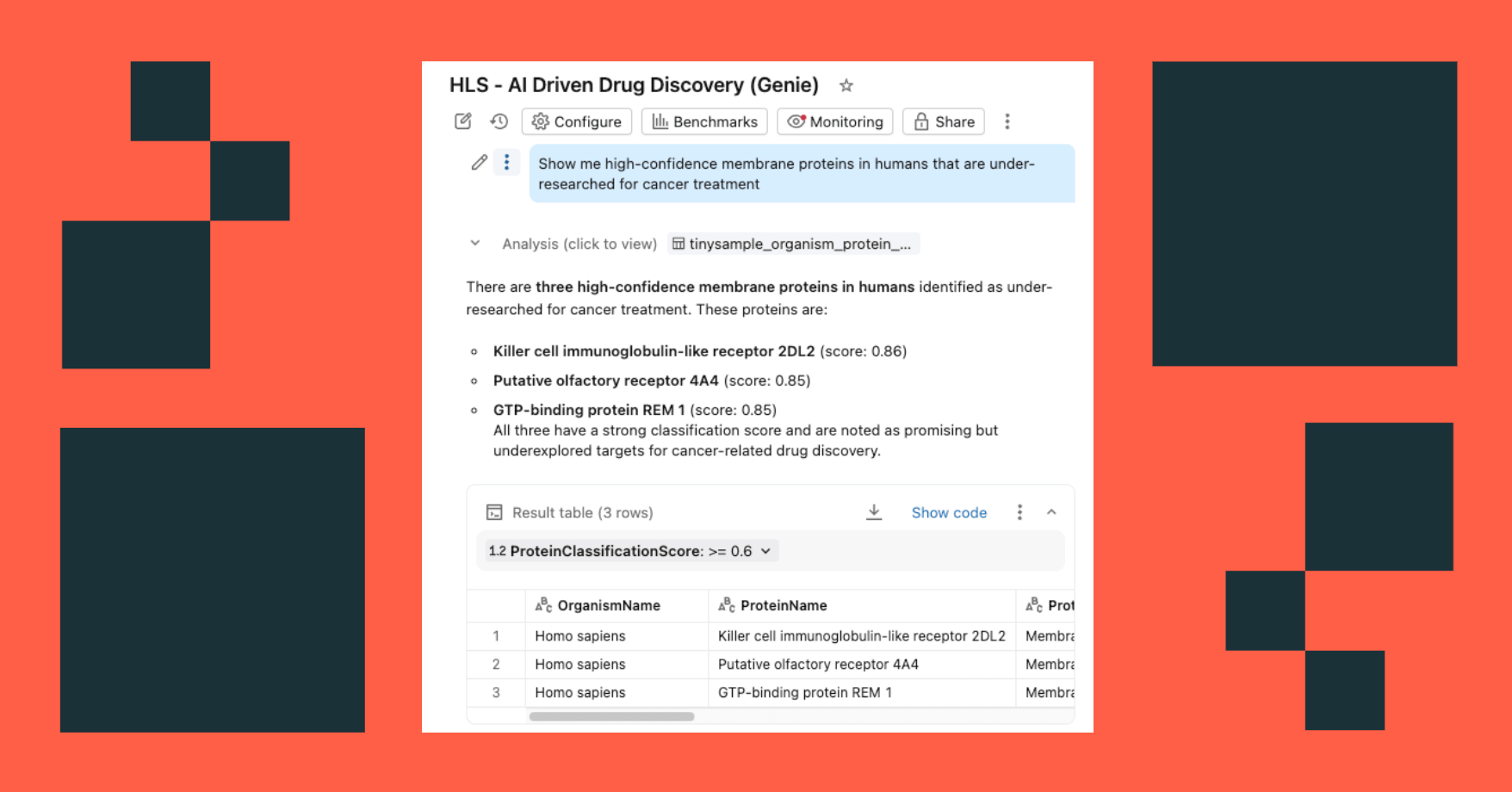
- Faça perguntas em linguagem natural: "Mostre-me proteínas de membrana de alta confiança em humanos que são pouco pesquisadas para o tratamento do câncer"
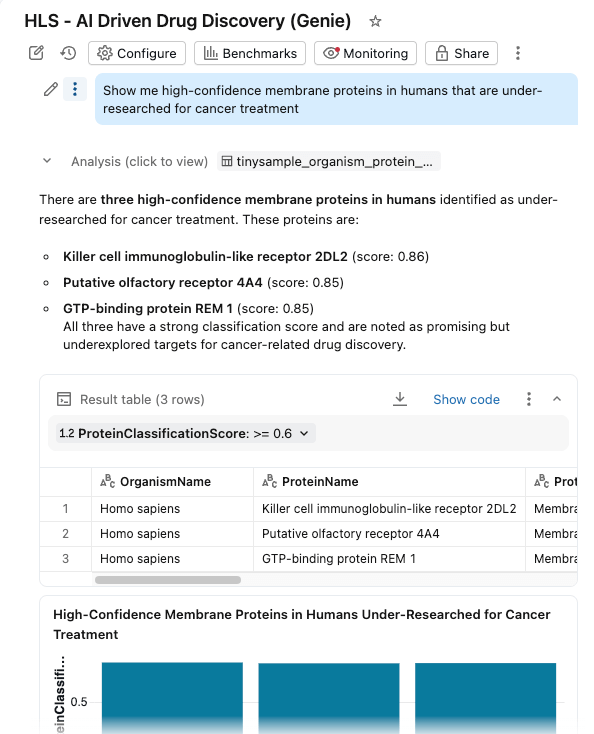
O dashboard consulta as mesmas tabelas governadas no Unity Catalog, com as AI Functions fornecendo enriquecimento sob demanda (ou processado em lotes).
A inteligência de dados transforma indústrias

O poder de uma plataforma unificada
O que torna esta solução atraente não se deve a um único componente — é o fato de que tudo roda em uma única plataforma:
| Capacidade | Recurso do Databricks |
|---|---|
| Ingestão de Dados e ETL | Pipelines Declarativos do LakeFlow |
| Governança de dados | Unity Catalog |
| Inferência de ML | GPU compute |
| Integração de LLM | FMAPI + Modelos Externos + AI Functions |
| Análises | Databricks SQL |
| Exploração | Painéis de AI/BI + AI/BI Genie Space |
Um ponto crucial é que não há movimentação de dados entre sistemas. Sem infraestrutura de MLOps separada. Sem ferramentas de BI desconectadas. A sequência de proteína que entra no pipeline flui por transformação, classificação, enriquecimento e acaba sendo consultável em linguagem natural — tudo dentro do mesmo ambiente governado.
O acelerador de soluções completo está disponível no GitHub:
github.com/databricks-industry-solutions/ai-driven-drug-discovery
Próximos passos
Este acelerador demonstra a arte do possível. Em produção, você pode estendê-lo para:
- Processar todo o banco de dados UniProt com endpoints de taxa de transferência provisionada
- Adicionar mais modelos de classificação (abertos ou personalizados) para diferentes propriedades de proteínas
- Crie pipelines de RAG sobre literatura científica para obter respostas de LLM mais fundamentadas
- Integrar com fluxos de trabalho de simulação molecular posteriores
- Conectar à previsão da estrutura de proteínas (AlphaFold/ESMFold) para adicionar contexto estrutural 3D às proteínas classificadas
- Estenda para outros formatos genômicos (FASTQ, VCF, BAM) usando o Glow para sequenciamento em grande escala e análise de variantes
A base está lá. A plataforma é unificada. O único limite é a ciência que você deseja acelerar. Comece hoje mesmo!
(Esta publicação no blog foi traduzida utilizando ferramentas baseadas em inteligência artificial) Publicação original
Nunca perca uma postagem da Databricks
Sign up
O que vem a seguir?

Saúde e ciências da vida
1 de dezembro de 2025/5 min de leitura
Databricks e NVIDIA: impulsionando a próxima geração de AI para as indústrias
Saúde e ciências da vida
21 de janeiro de 2026/14 min de leitura