Desidentificando imagens médicas com bom custo-benefício com modelos de linguagem de visão no Databricks
Os VLMs são surpreendentemente bons em desidentificar imagens médicas e têm bom custo-benefício no Databricks.
Summary
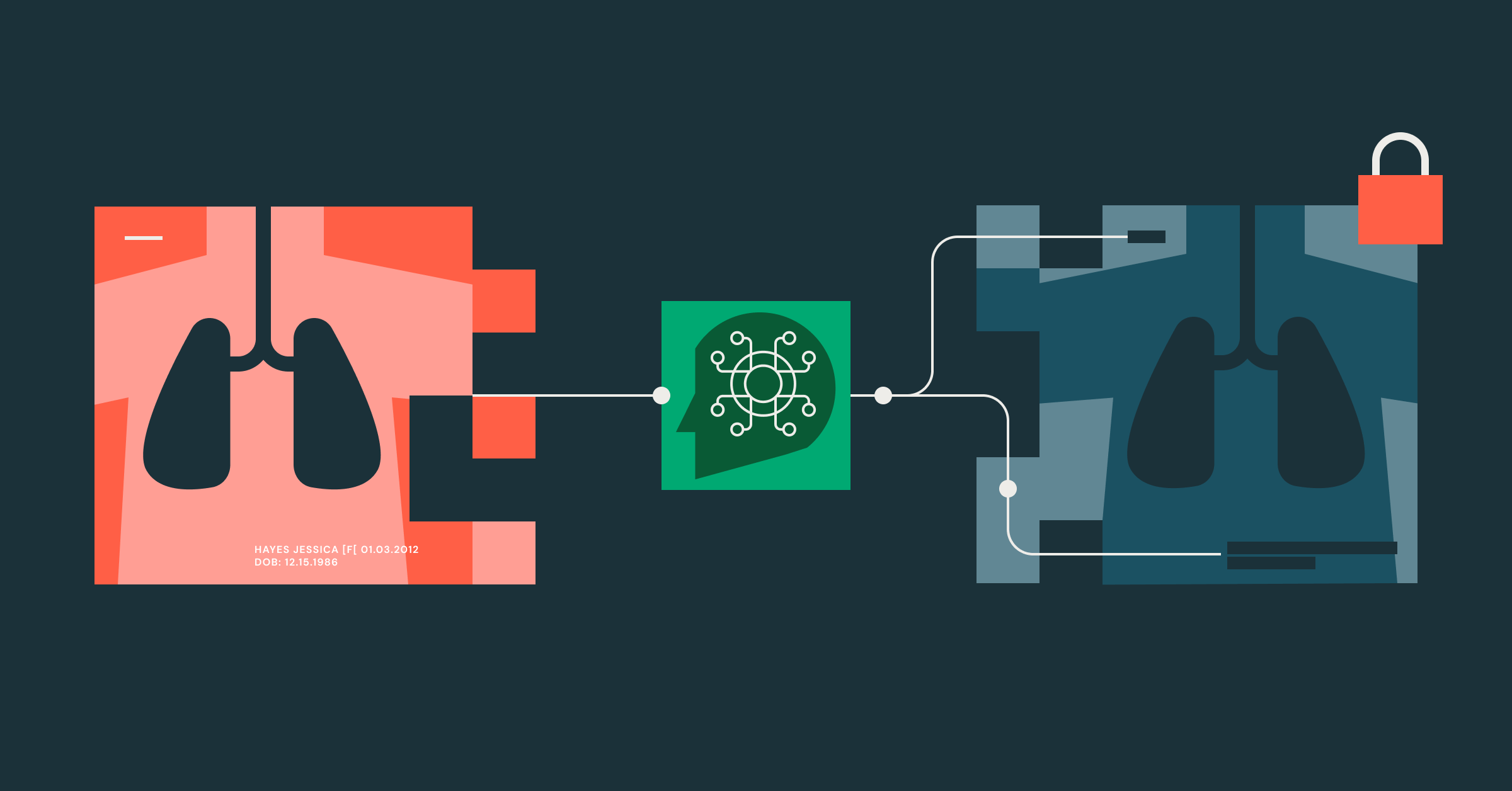
- Problema de negócio - Para tornar os prontuários médicos seguros e úteis para a pesquisa clínica, e principalmente para proteger contra a quebra de cegamento de um ensaio clínico, eles precisam ser desidentificados. Embora a desidentificação de texto seja um problema bem resolvido, a desidentificação de imagens ainda deixa a desejar. Acreditamos que os VLMs avançaram a ponto de se tornarem uma solução viável para a desidentificação de imagens em grande escala.
- Soluções testadas - Durante este projeto, testamos o Presidio, uma solução de referência de código aberto, e VLMs comerciais e de código aberto (Modelos de Linguagem Visual)
- Resultados - Em nossos experimentos, os VLMs são surpreendentemente bons em detectar PHI em uma imagem, com um recall de 100%. Além do desempenho superior, os VLMs têm um bom custo-benefício e são fáceis de usar com poucos ajustes, em comparação com as abordagens clássicas de OCR e somente texto.
Por que a necessidade de desidentificação escalável de imagens
Imagens médicas, como raios-X e MRIs, além de ajudar no diagnóstico, planejamento de tratamento e monitoramento de doenças, são cada vez mais usadas para além do cuidado individual do paciente, a fim de embasar pesquisas médicas mais amplas, políticas de saúde pública e o desenvolvimento de novas ferramentas de diagnóstico baseadas em AI. Este uso secundário de prontuários médicos, apesar de extremamente benéfico, precisa passar pela desidentificação de informações de saúde protegidas (PHI) para proteger a privacidade do paciente e estar em conformidade com regulamentações como a HIPAA.
A escala crescente dos datasets de imagens médicas exige métodos de desidentificação confiáveis e eficientes, garantindo que as imagens possam ser usadas com segurança e ética para o avanço da ciência médica. Para esse fim, apresentamos o Pixels Solution Accelerator com um Spark ML pipelines que utiliza modelos de linguagem de visão (VLM) em paralelo para desidentificar imagens médicas no formato amplamente utilizado, digital Imaging and Communications in Medicine (DICOM).
Um arquivo DICOM contém imagens e metadados de texto (leia mais aqui). Aqui, focamos em nosso novo recurso de desidentificação de imagens. Vale ressaltar que o Pixels, nosso kit de ferramentas DICOM, também desidentifica metadados, além de oferecer ingestão e segmentação escaláveis de DICOM, tudo em um aplicativo da web.
Como desidentificar PHI gravadas em imagens DICOM
Após instalar o pacote python Pixels, faça a execução do DicomPhiPipeline da seguinte forma:
Lê o caminho de um arquivo DICOM em uma coluna em um dataframe do Spark e gera 2 colunas:
- uma resposta de um VLM (especificado no endpoint)
- um caminho de arquivo DICOM com PHI mascarada
Como parte do DicomPhiPipeline, a redação é realizada usando o EasyOCR. A redação pode ser realizada independentemente da detecção de PHI do VLM (redact_even_if_undetected=True) ou realizada condicionalmente à detecção de PHI do VLM (redact_even_if_undetected=False). Recomendamos a última opção, pois o EasyOCR tem a tendência de ocultar em excesso dados que não são PHI. Ao usar como condição as imagens que o VLM detectou como positivas para PHI, o EasyOCR terá menor probabilidade de redigir as imagens sem PHI.
Comparação com outros métodos de detecção de PHI
A competição
Testamos o pipeline de detecção de PHI em imagens da Pixels com um fornecedor comercial e uma solução de código aberto amplamente utilizada, a Presidio. Tanto o fornecedor quanto a Presidio usaram o OCR para primeiro extrair o texto das imagens e depois aplicar um modelo de linguagem para classificar se o texto era PHI ou não. O OCR integrado também segmentou o texto sensível e aplicou uma máscara de preenchimento dentro daquelas caixas delimitadoras.
Além disso, comparamos vários VLMs: GPT-4o, Claude 3.7 Sonnet e o Llama 4 Maverick de código aberto.
Conjuntos de dados
A comparação foi feita em datasets DICOM públicos, MIDI-B, nos quais fizemos a subamostragem para 70 imagens para criar um dataset balanceado com um número aproximadamente igual de imagens com e sem PHI.
Resultados
| Tarefa: detecção de PHI em imagens DICOM | MIDI-B (70) | ||||
|---|---|---|---|---|---|
| Solução | Estimativas de Custo por 100 mil imagens | Recall | Precisão | Especificidade | NPV |
| ISV (comercial) | $4.400 por mês pré-pago | 1,0 | 0,71 | 0.93 | 1,0 |
| Presidio (OSS) | $0 | 0.7 | 0.7 | 0.95 | 0.95 |
| Claude 3.7 Sonnet | $270 | 1,0 | 1,0 | 1,0 | 1,0 |
| GPT-4o | $150 | 1,0 | 1,0 | 1,0 | 1,0 |
| Llama 4 Maverick (OSS) | $45 | 1,0 | 0,91 | 0,98 | 1,0 |
Tanto o Claude 3.7 Sonnet quanto o GPT-4o tiveram um desempenho perfeito na detecção de PHI. O Llama 4 Maverick teve 100% de recall, mas 91% de precisão, pois às vezes identifica incorretamente como PHI textos na imagem que não são PHI. No entanto, o Llama 4-Maverick ainda oferece um bom desempenho, especialmente para usuários que preferem redigir em excesso para evitar a omissão de qualquer PHI. Nesse caso, ele tem uma taxa de omissão falsa de PHI igual a zero (ou seja, NPV próximo de 1) e revocação de 1, portanto, pode ser um bom equilíbrio entre desempenho e custo.
Em nossos testes, usamos o Presidio e a solução comercial prontos para uso, com as configurações default. Observamos que o desempenho, tanto em termos de acurácia quanto de velocidade, dependia muito da escolha do OCR. É provável que o desempenho deles possa ser melhorado com alternativas como o Azure Document Intelligence.
Por que funciona
Pesquisamos a literatura sobre a desidentificação de texto gravado em imagens médicas e aprendemos com o sucesso relatado do uso de OCR, LLMs (por exemplo, BERT, Bi-LSTM, GPT) e/ou VLMs. Nossa decisão de usar VLM para detectar PHI e EasyOCR para detectar caixas delimitadoras de texto foi guiada pelo sucesso relatado por Truong et al. 2025.
Os VLMs substituem o OCR tradicional, que tem um reconhecimento de texto ruim e geralmente introduz erros de digitação.
Na maioria dos métodos de desidentificação relatados, o OCR foi frequentemente usado como o primeiro passo para extrair texto de imagens inseridas em um LLM. No entanto, observamos que ferramentas de OCR como o tessaract e o EasyOCR eram geralmente ruins e lentas no reconhecimento de texto (ou seja, leitura), muitas vezes lendo incorretamente certos caracteres e introduzindo erros de digitação inadvertidamente e comprometendo a detecção de PHI downstream. Para mitigar isso, usamos um VLM para ler o texto embutido na imagem e classificar se o texto era PHI; os VLMs foram surpreendentemente bons nisso.
EasyOCR para detectar caixas delimitadoras para redação quando os VLMs não conseguem alterar imagens
No entanto, os VLMs não conseguem produzir imagens com dados ocultos. Assim, usamos o OCR para fazer o que ele faz de melhor, ou seja, detectar texto, para fornecer as coordenadas da caixa delimitadora para o mascaramento subsequente. É importante notar que, embora tenha havido tentativas recentes de fazer o ajuste fino de um VLM para gerar coordenadas de caixas delimitadoras Chen et al. 2025, optamos por uma solução mais simples, montada com ferramentas prontas para uso (VLM, EasyOCR).
Paralelismo do Spark para escalabilidade de nível de produção
Embora o Databricks tivesse um recurso de inferência em lote com LLMs (ai_functions), atualmente ele não oferece suporte para VLMs. Dessa forma, implementamos uma versão escalável para VLM e EasyOCR usando Pandas UDF. Trabalhando com um grande cliente farmacêutico, o paralelismo do Spark acelerou o processo de desidentificação de 105 minutos para 6 minutos em uma execução de teste de 1.000 quadros DICOM! Ao ampliar para a carga de trabalho completa de 100.000 quadros DICOM, a aceleração e a economia de custos foram significativas.
Resumo
Dado o poder, a facilidade e a economia dos VLMs, conforme demonstrado pelos complementos do acelerador de soluções Pixels 2.0, não é apenas viável, mas também prudente proteger seus estudos clínicos críticos e estudos de imagem relacionados com detecção de PHI escalável.
Embora o Pixels tenha sido projetado para arquivos DICOM, descobrimos que nossos clientes o adaptam para outros formatos de imagem, como JPEG, Whole Slide Images, SVS e assim por diante.
As atualizações são postadas em nosso repo do github, então este é um bom momento para atualizar ou experimentar o acelerador de soluções Databricks Pixels 2.0. Entre em contato com sua equipe de account da Databricks para discutir o processamento de seus dados de imagem e casos de uso de AI/ML. Os autores ficariam felizes em receber seu contato pelo LinkedIn, caso ainda não nos conheçamos.
(This blog post has been translated using AI-powered tools) Original Post
Acompanhe-nos
Artigos relacionados

Insights
August 7, 2025/7 min de leitura
Ingestão de Imagens Médicas 7x Mais Rápida com a API Python Data Source
Nunca perca uma postagem da Databricks
Sign up
O que vem a seguir?

Saúde e ciências da vida
December 1, 2025/5 min de leitura
Databricks e NVIDIA: impulsionando a próxima geração de AI para as indústrias
Mosaic Research
December 1, 2025/18 min de leitura