Aceleración del descubrimiento de fármacos: de archivos FASTA a información de GenAI en Databricks
Cómo construir un pipeline de extremo a extremo que combine ingeniería de datos, modelos de lenguaje de proteínas y GenAI en la plataforma Databricks.
Publicado: 4 de febrero de 2026
por Ram Goli y May Merkle-Tan
Summary
- Procese datos biológicos a escala con las canalizaciones declarativas de Lakeflow para transformar secuencias de proteínas FASTA sin procesar en tablas listas para el análisis en Unity Catalog.
- Clasificar proteínas con modelos de transformador aprovechando ProtBERT, un modelo de lenguaje de proteínas, para identificar proteínas de transporte de membrana: objetivos farmacológicos clave.
- Consulte información sobre proteínas en lenguaje natural a través de funciones de IA que conectan los LLM directamente a sus datos, lo que permite a los investigadores explorar candidatos a fármacos prometedores de forma conversacional.
El desarrollo de fármacos es conocido por ser lento y costoso. El ciclo de vida promedio de la investigación y el desarrollo (I+D) dura entre 10 y 15 años, y una parte significativa de los candidatos fracasa durante los ensayos clínicos. Un cuello de botella importante ha sido la identificación de las proteínas diana adecuadas en una fase temprana del proceso.
Las proteínas son las "moléculas de trabajo" de los organismos vivos: catalizan reacciones, transportan moléculas y actúan como los blancos de la mayoría de los fármacos modernos. La capacidad de clasificar rápidamente las proteínas, comprender sus propiedades e identificar candidatos poco investigados podría acelerar drásticamente el proceso de descubrimiento (p. ej., Wozniak et al., 2024, Nature Chemical Biology).
Aquí es donde la convergencia de la ingeniería de datos, el machine learning (ML) y la IA generativa se vuelve transformadora. De hecho, puede construir todo este pipeline en una única plataforma: la Databricks Data Intelligence Platform.
Lo que estamos construyendo
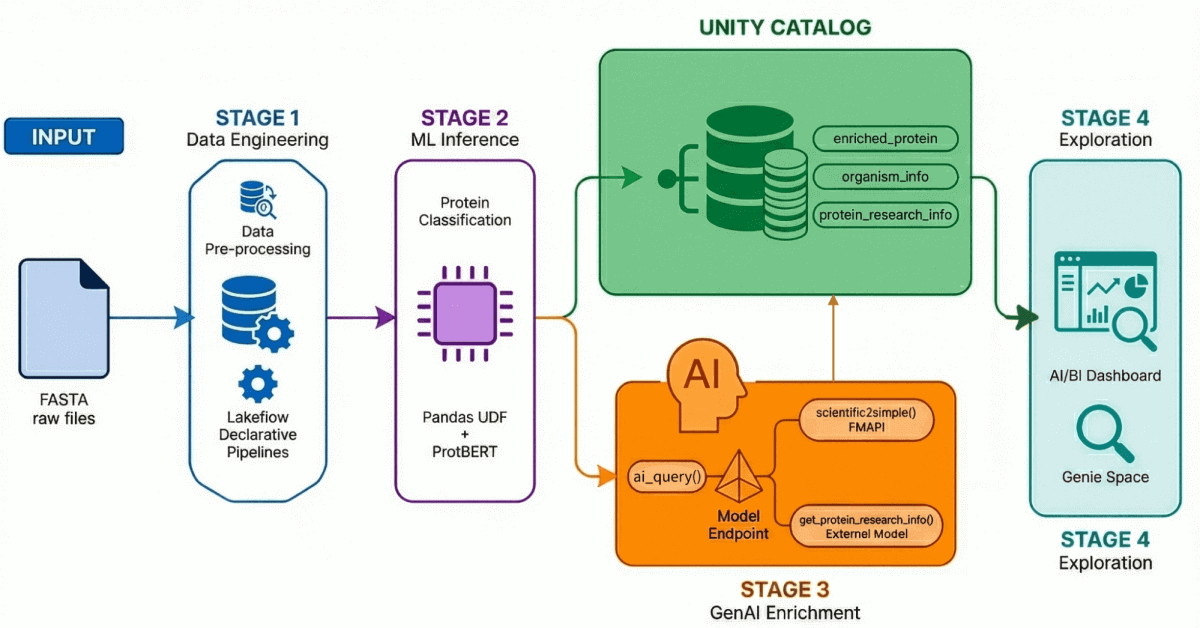
Nuestro acelerador de soluciones para el descubrimiento de fármacos impulsado por IA demuestra un flujo de trabajo de extremo a extremo a través de cuatro procesos clave:
- Ingesta y procesamiento de datos: Se ingieren y procesan más de 500 000 secuencias de proteínas de UniProt.
- Clasificación impulsada por IA: se utiliza un modelo transformer para clasificar estas proteínas como hidrosolubles o de transporte de membrana.
- Generación de información: los datos de proteínas se enriquecen con información de investigación generada por LLM.
- Exploración en lenguaje natural: todos los datos procesados y enriquecidos están accesibles a través de un dashboard y un entorno con IA que admite consultas en lenguaje natural.
Repasemos cada etapa:
Etapa 1: Ingeniería de datos con canalizaciones declarativas de Lakeflow
Los datos biológicos en bruto rara vez llegan en un formato limpio y listo para el análisis. Nuestros datos de origen vienen en archivos FASTA, un formato estándar para representar secuencias de proteínas que se ve más o menos así:
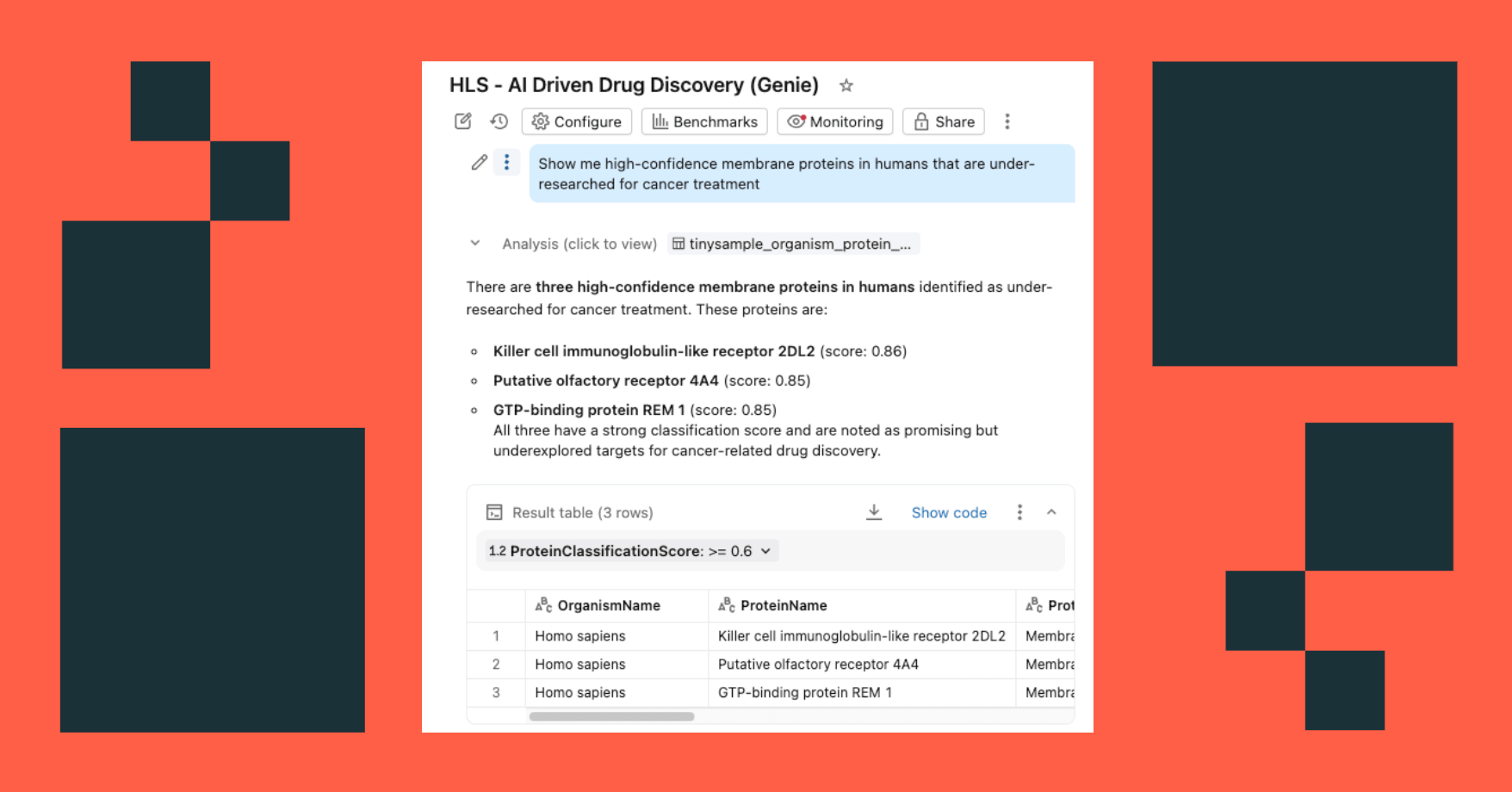
Para un ojo inexperto, estos datos de secuencia son casi imposibles de interpretar: una densa cadena de códigos de aminoácidos de una sola letra. Sin embargo, al final de este proceso, los investigadores pueden consultar estos mismos datos en lenguaje natural y hacer preguntas como "Muéstrame proteínas de membrana poco investigadas en humanos con una alta confianza de clasificación" para recibir información útil a cambio.
Mediante el uso de Lakeflow Declarative Pipelines, construimos una arquitectura medallion que refina progresivamente estos datos:
- Capa Bronze: ingesta sin procesar de archivos FASTA con BioPython, extrayendo ID y secuencias.
- Capa Silver: Análisis y estructuración—extraemos nombres de proteínas, información de organismos, nombres de genes y otros metadatos mediante transformaciones con regex.
- Capa Gold/Enriquecida: datos seleccionados y listos para el análisis, enriquecidos con métricas derivadas como el peso molecular, para dashboards, modelos de ML e investigación posterior. Esta es la capa de confianza que los analistas y científicos consultan directamente.
El resultado: datos de proteínas limpios y gobernados en Unity Catalog, listos para el ML y el análisis posteriores. Es fundamental destacar que el linaje de datos que se extiende más allá de esta etapa hacia las otras etapas (resaltadas a continuación) aporta un valor increíble para la reproducibilidad científica.
Etapa 2: Clasificación de proteínas con modelos Transformer
No todas las proteínas son iguales cuando se trata del descubrimiento de fármacos. Las proteínas de transporte de membrana —aquellas incrustadas en las membranas celulares— son objetivos farmacológicos particularmente importantes porque controlan lo que entra y sale de las células.
Utilizamos ProtBERT-BFD, un modelo de lenguaje de proteínas basado en BERT de Rostlab, con un ajuste fino específico para la clasificación de proteínas de membrana. Este modelo trata las secuencias de aminoácidos como un lenguaje, aprendiendo las relaciones contextuales entre los residuos para predecir la función de las proteínas.
El modelo genera como resultado una clasificación (como de membrana o soluble) junto con una puntuación de confianza, que volvemos a escribir en Unity Catalog para el filtrado y análisis posteriores.
Etapa 3: Enriquecimiento de datos con GenAI
La clasificación nos dice qué es una proteína. Pero los investigadores necesitan saber por qué es importante: ¿cuál es la investigación reciente? ¿Dónde están las lagunas? ¿Es este un objetivo farmacológico poco explorado?
Aquí es donde entran en juego los LLM. Aprovechando tanto la API de modelos fundacionales de Databricks como los endpoints de modelos externos, creamos funciones de IA registradas que enriquecen los registros de proteínas con contexto de investigación.
Etapa 4: Exploración en lenguaje natural
Integramos todo en un panel de IA/BI con Genie Space habilitado.
Los investigadores ahora pueden:
- Filtrar proteínas por organismo, puntuación de clasificación y tipo de proteína
- Explorar distribuciones de pesos moleculares y confianza de la clasificación
- Haga preguntas en lenguaje natural: "Muéstrame las proteínas de membrana de alta confianza en humanos que están poco investigadas para el tratamiento del cáncer"
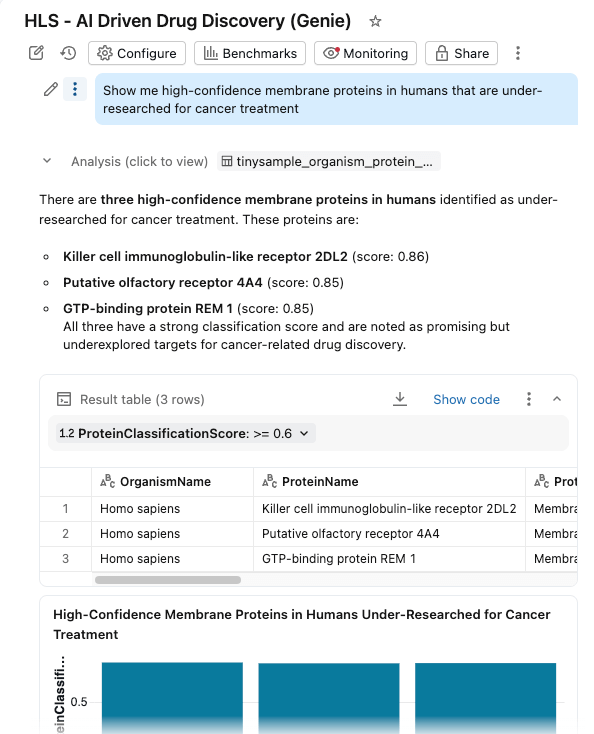
El dashboard consulta las mismas tablas gobernadas en Unity Catalog, con AI Functions que proporcionan enriquecimiento on-demand (o procesado por lotes).
La inteligencia de datos remodela las industrias

El poder de una plataforma unificada
Lo que hace que esta solución sea atractiva no se debe a un único componente, sino a que todo se ejecuta en una sola plataforma:
| Capacidad | Característica de Databricks |
|---|---|
| Ingesta de datos & ETL | Pipelines declarativos de Lakeflow |
| Gobernanza de datos | Unity Catalog |
| Inferencia de ML | GPU Compute |
| Integración de LLM | FMAPI + Modelos externos + Funciones de IA |
| Analítica | Databricks SQL |
| Exploración | Dashboards de AI/BI + Espacio Genie de AI/BI |
Un punto crítico es que no hay movimiento de datos entre los sistemas. Sin infraestructura de MLOps separada. Sin herramientas de BI desconectadas. La secuencia de proteína que ingresa al pipeline fluye a través de la transformación, la clasificación y el enriquecimiento, y termina siendo consultable en lenguaje natural, todo dentro del mismo entorno gobernado.
El acelerador de soluciones completo está disponible en GitHub:
github.com/databricks-industry-solutions/ai-driven-drug-discovery
¿Qué sigue?
Este acelerador demuestra el arte de lo posible. En producción, podría ampliarlo para:
- Procese toda la base de datos de UniProt con terminales de rendimiento aprovisionado
- Agregar más modelos de clasificación (abiertos o personalizados) para diferentes propiedades de las proteínas
- Crea RAG pipelines sobre bibliografía científica para obtener respuestas de LLM más fundamentadas
- Integración con flujos de trabajo de simulación molecular posteriores
- Conéctese a la predicción de la estructura de las proteínas (AlphaFold/ESMFold) para agregar un contexto estructural en 3D a las proteínas clasificadas
- Extiende a otros formatos genómicos (FASTQ, VCF, BAM) usando Glow para la secuenciación a gran escala y el análisis de variantes
Las bases están sentadas. La plataforma está unificada. El único límite es la ciencia que quieres acelerar. ¡Comienza hoy mismo!
(Esta entrada del blog ha sido traducida utilizando herramientas basadas en inteligencia artificial) Publicación original
No te pierdas ninguna publicación de Databricks.
Sign up
¿Qué sigue?

Saúde e ciências da vida
1 de diciembre de 2025/5 min de lectura
Databricks e NVIDIA: impulsionando a próxima geração de AI para as indústrias
Saúde e ciências da vida
21 de enero de 2026/14 min de lectura