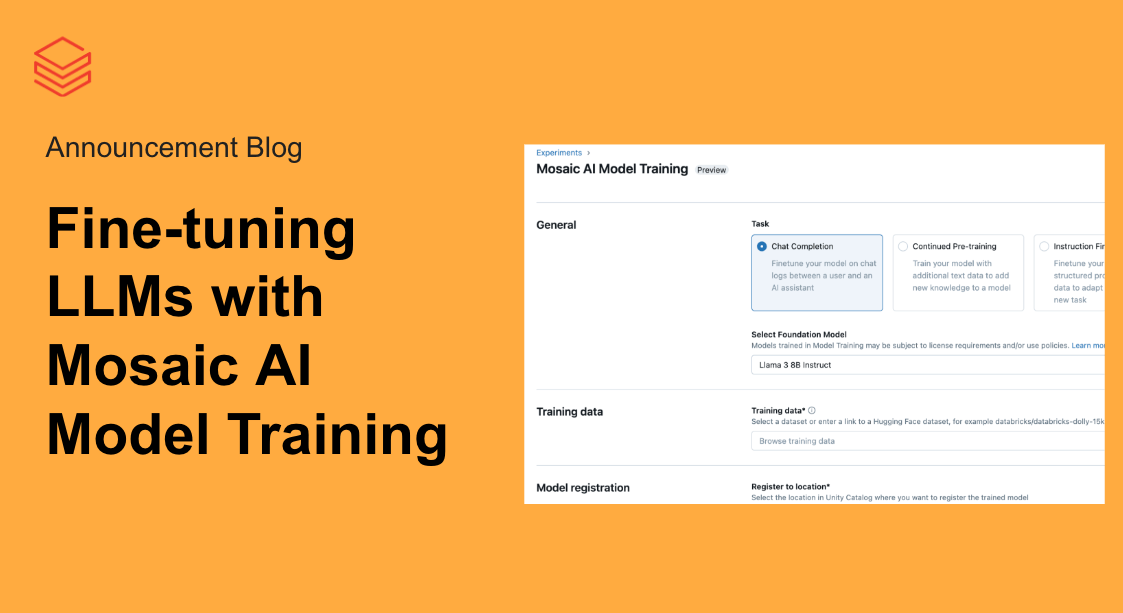
Databricks Delta를 사용하여 대규모 유전체학 파이프라인 간소화

Delta Lake 사용을 시작하는 데 필요한 단계별 안내를 담은 오라일리의 새 ebook을 미리 만나보세요.
Databricks에서 이 노트북을 사용해 보세요
이 블로그는 '대규모 유전체학 분석' 시리즈의 첫 번째 블로그입니다. 이 시리즈에서는 고객이 Databricks Unified Analytics Platform for Genomics 을 사용하여 인구 규모의 유전체 데이터를 분석하는 방법을 시연합니다. 유전체학 파이프라인의 출력부터 시작하여, 이 시리즈는 Databricks를 사용하여 샘플 품질 관리, 공동 유전자형 분석, 코호트 품질 관리, 고급 통계 유전학 분석을 실행하는 방법에 대한 튜토리얼을 제공합니다.
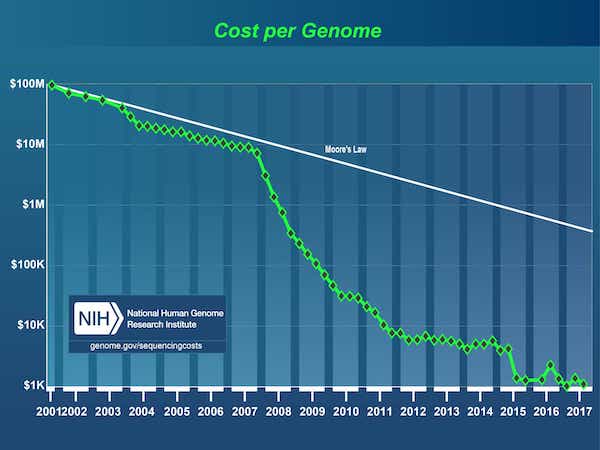
2003년 인간 게놈 프로젝트 가 완료된 이후 DNA 시퀀싱 비용이 최초 게놈의 경우 30억 달러1 에서 현재 1,000달러 미만으로 급격히 떨어지면서 데이터가 폭발적으로 증가했습니다.
[1] 인간 게놈 프로젝트 는 에너지부와 국립보건원이 주도한 30억 달러 규모의 프로젝트로, 1990년에 시작되어 2003년에 완료되었습니다.
출처: DNA 시퀀싱 비용: 데이터
결과적으로, 유전체학 분야는 이제 기업들이 인구 규모로 DNA 시퀀싱을 시작하는 단계까지 성숙했습니다. 하지만 DNA 코드를 시퀀싱하는 것은 첫 번째 단계에 불과하며, 이후 가공되지 않은 데이터를 분석에 적합한 형식으로 변환해야 합니다. 일반적으로 이는 일련의 생물정보학 도구를 사용자 지정 스크립트와 함께 연결하고, 단일 노드에서 한 번에 하나의 샘플씩 데이터를 처리하여 최종적으로 유전체 변이 모음을 얻는 방식으로 수행됩니다. 오늘날 생물정보학 과학자들은 대부분의 시간을 이러한 파이프라인을 구축하고 유지 관리하는 데 사용합니다. 유전체 데이터 세트가 페타바이트 규모로 확장됨에 따라 다음과 같은 간단한 질문에도 시기적절하게 답하기가 어려워졌습니다.
- 이번 달에 몇 개의 샘플을 시퀀싱했나요?
- 총 몇 개의 고유한 변이가 검출되었나요?
- 다양한 변이 클래스에서 얼마나 많은 변이가 발견되었습니까?
이 문제를 더욱 악화시키는 것은, 수천 명의 개인 데이터를 저장, 추적, 버전 관리하면서 동시에 액세스 및 쿼리가 가능하도록 유지할 수 없다는 점입니다. 결과적으로 연구원들은 분석을 수행할 때 게놈 데이터의 하위 집합을 복제하는 경우가 많아 전체 스토리지 공간과 비용이 증가하게 됩니다. 이 문제를 완화하기 위해 오늘날 연구자들은 일반적으로 6개월에서 2년 사이의 '데이터 동결' 전략을 사용하는데, 이 기간 동안 새로운 데이터에 대한 작업을 중단하고 대신 기존 데이터의 동결된 복사본에 집중합니다. 더 짧은 기간에 걸쳐 점진적으로 분석을 구축할 수 있는 솔루션이 없어 연구 진행이 더뎌집니다.
산업적 규모의 유전체 데이터를 처리하는 동시에 과학자들이 데이터를 탐색하고 분석 파이프라인을 반복적으로 개선하며 새로운 인사이트를 도출할 수 있는 유연성을 유지하는 강력한 소프트웨어에 대한 필요성이 절실합니다.
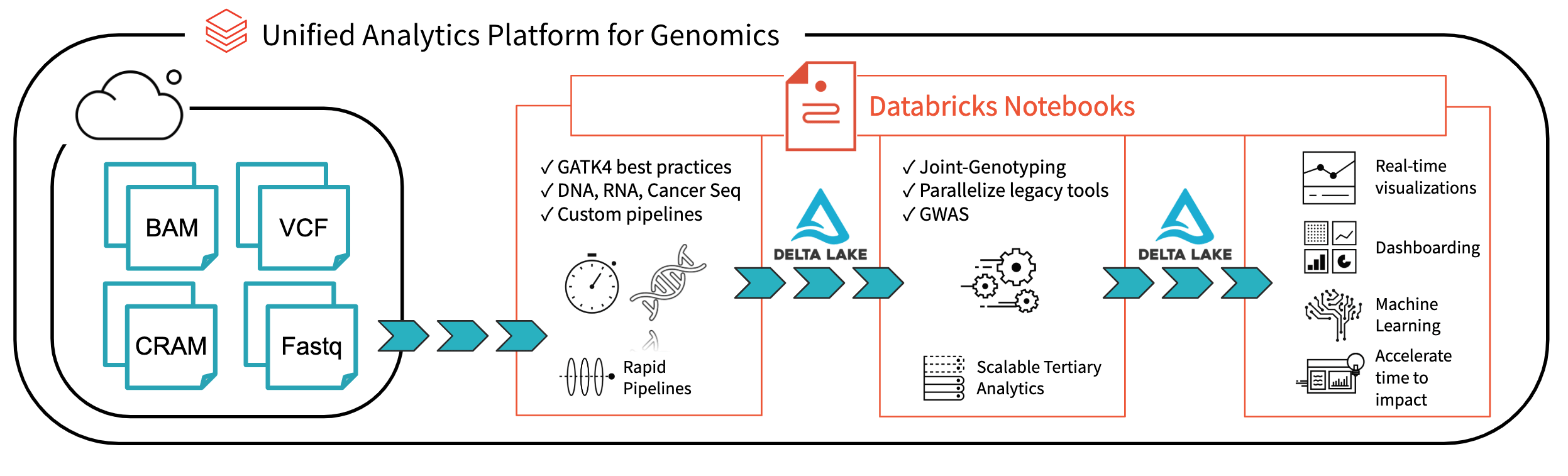
그림 1. Databricks를 사용한 엔드투엔드 유전체학 분석 아키텍처
Databricks Delta: 실시간 빅데이터 분석을 위한 통합 관리 시스템을 통해 Databricks 플랫폼은 오늘날 연구자들이 직면한 데이터 거버넌스, 데이터 액세스 및 데이터 분석 문제를 해결하는 데 큰 진전을 이루었습니다. Databricks Delta Lake를 사용하면 모든 유전체학 데이터를 한곳에 저장하고 새 데이터가 수집될 때 실시간으로 업데이트되는 분석을 생성할 수 있습니다. 유전체학 파일 형식의 읽기, 쓰기, 처리를 위한 유전체학용 Unified Analytics Platform (UAP4G)의 최적화와 결합하여 유전체학 파이프라인 워크플로를 위한 엔드투엔드 솔루션을 제공합니다. UAP4G 아키텍처는 유연성을 제공하여 고객이 자체 파이프라인을 연결하고 자체 3차 분석을 개발할 수 있도록 합니다. 예를 들어, 자동화된 방식으로 계산 및 표시되고 특정 요구 사항에 맞게 사용자 정의할 수 있는 품질 관리 지표 및 시각화를 보여주는 다음 대시보드를 소개합니다.
https://www.youtube.com/watch?v=73fMhDKXykU
이 블로그의 나머지 부분에서는 샘플 처리가 완료될 때 실시간으로 업데이트되는 위의 품질 관리 대시보드를 구축하기 위해 저희가 수행한 단계를 살펴보겠습니다. 유전체 데이터 처리를 위해 Delta 기반 파이프라인을 사용함으로써 고객은 이제 실시간으로 샘플별 가시성을 제공하는 방식으로 파이프라인을 운영할 수 있습니다. Databricks 노트북(및 GitHub, MLflow와 같은 통합 기능)을 통해 결과의 재현성을 보장하는 방식으로 분석을 추적하고 버전을 관리할 수 있습니다. 생물정보학자들은 파이프라인 유지 관리에 드는 시간을 줄이고 새로운 발견에 더 많은 시간을 할애할 수 있습니다. 저희는 UAP4G가 임시 분석에서 산업 규모의 프로덕션 유전체학으로의 전환을 이끄는 엔진이 되어, 유전학과 질병 간의 link에 대한 더 나은 인사이트를 제공할 것으로 보고 있습니다.
샘플 데이터 읽기
소규모 샘플 코호트의 변이 데이터를 읽는 것으로 시작하겠습니다. 다음 구문은 특정 `sampleId`의 데이터를 읽어 Databricks Delta 형식으로 `delta_stream_output` 폴더에 저장합니다.
참고: annotations_etl_parquet 폴더에는 1000 genomes 데이터 세트 에서 생성된 주석이 Parquet 형식으로 저장되어 있습니다. 이러한 주석의 ETL 및 처리는 Databricks의 유전체학 Unified Analytics Platform을 사용하여 수행되었습니다.
Start Databricks Delta 테이블 스트리밍
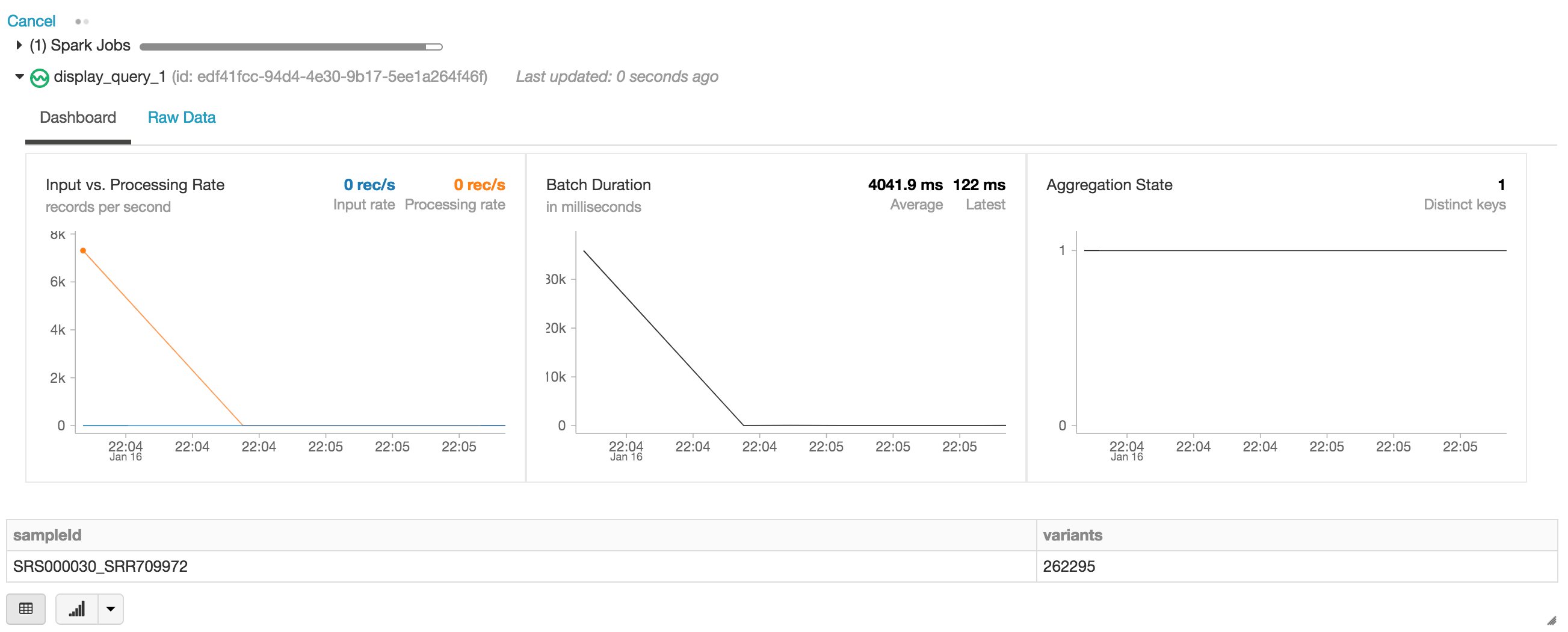
다음 문에서는 Databricks Delta 형식을 사용하여 데이터 stream(readStream 경유)을 읽는 exomes Apache Spark DataFrame을 생성합니다. 이것은 계속 실행되는 동적 DataFrame입니다. 즉, 데이터가 delta_stream_output 폴더에 기록될 때 exomes DataFrame은 새 데이터를 로드합니다. exomes DataFrame을 보려면 DataFrame 쿼리를 실행하여 sampleId별로 그룹화된 변이 개수를 찾을 수 있습니다.
display 문을 실행하면 Databricks 노트북이 스트리밍 작업을 모니터링하기 위한 스트리밍 대시보드를 제공합니다. 스트리밍 작업 바로 아래에 display 문의 결과(즉, sample_id별 변형 수)가 있습니다.
엑솜 데이터프레임을 기반으로 다른 데이터프레임 쿼리를 실행하여 초기 질문 세트에 대한 답을 계속 찾아보겠습니다.
단일 염기 변이 수
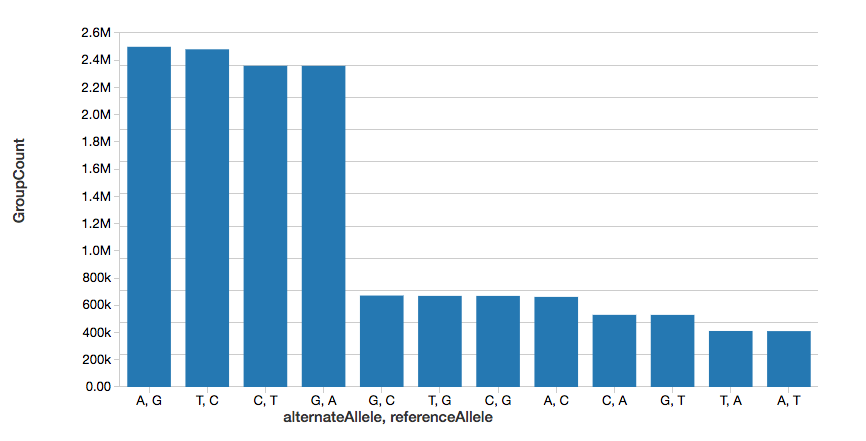
예제를 계속해서 다음 그래프에 표시된 것처럼 단일 염기 변이(SNV)의 수를 빠르게 계산할 수 있습니다.
참고: display 명령어는 Databricks 워크스페이스의 일부로서 Databricks 시각화를 사용하여 DataFrame을 볼 수 있도록 합니다(즉, 코딩 필요 없음).
변이 수
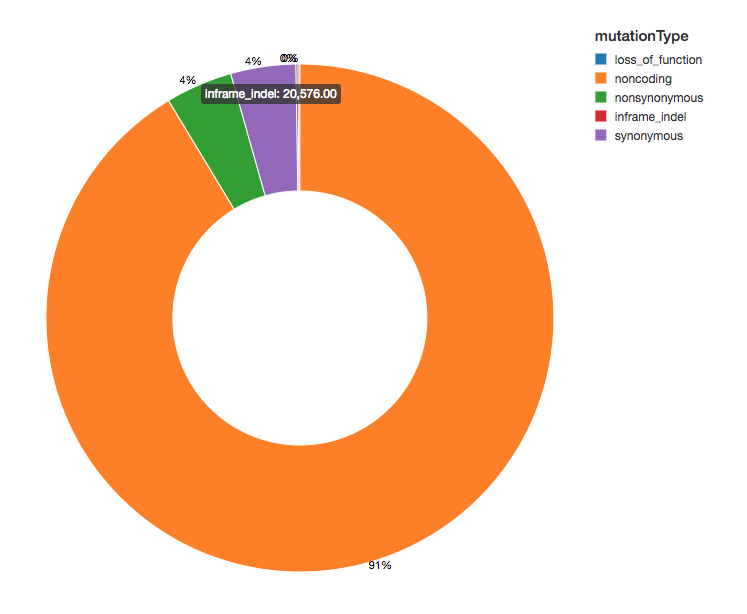
변이에 기능적 효과 주석을 달았으므로, 우리가 보는 변이 효과의 분포를 살펴봄으로써 분석을 계속할 수 있습니다. 검출된 변이의 대부분은 단백질을 코딩하는 영역에 인접하며, 이를 비코딩 변이(noncoding variants)라고 합니다.
ETL 시작하기

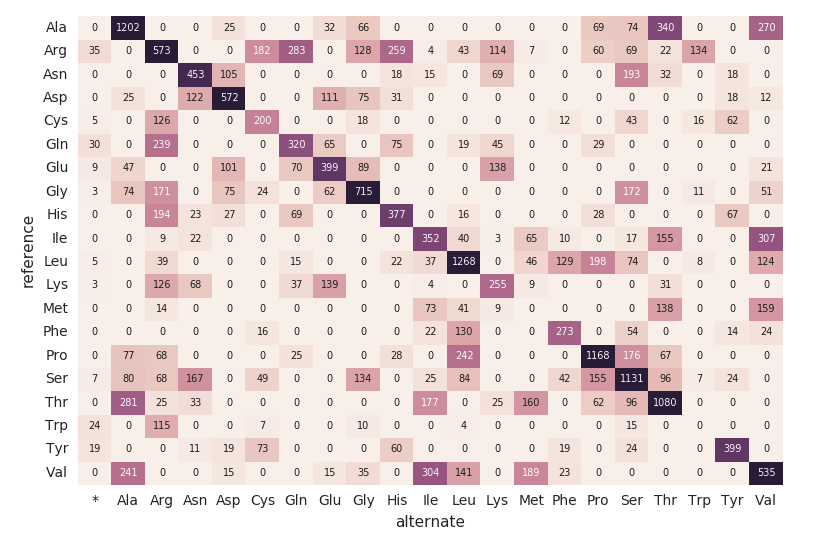
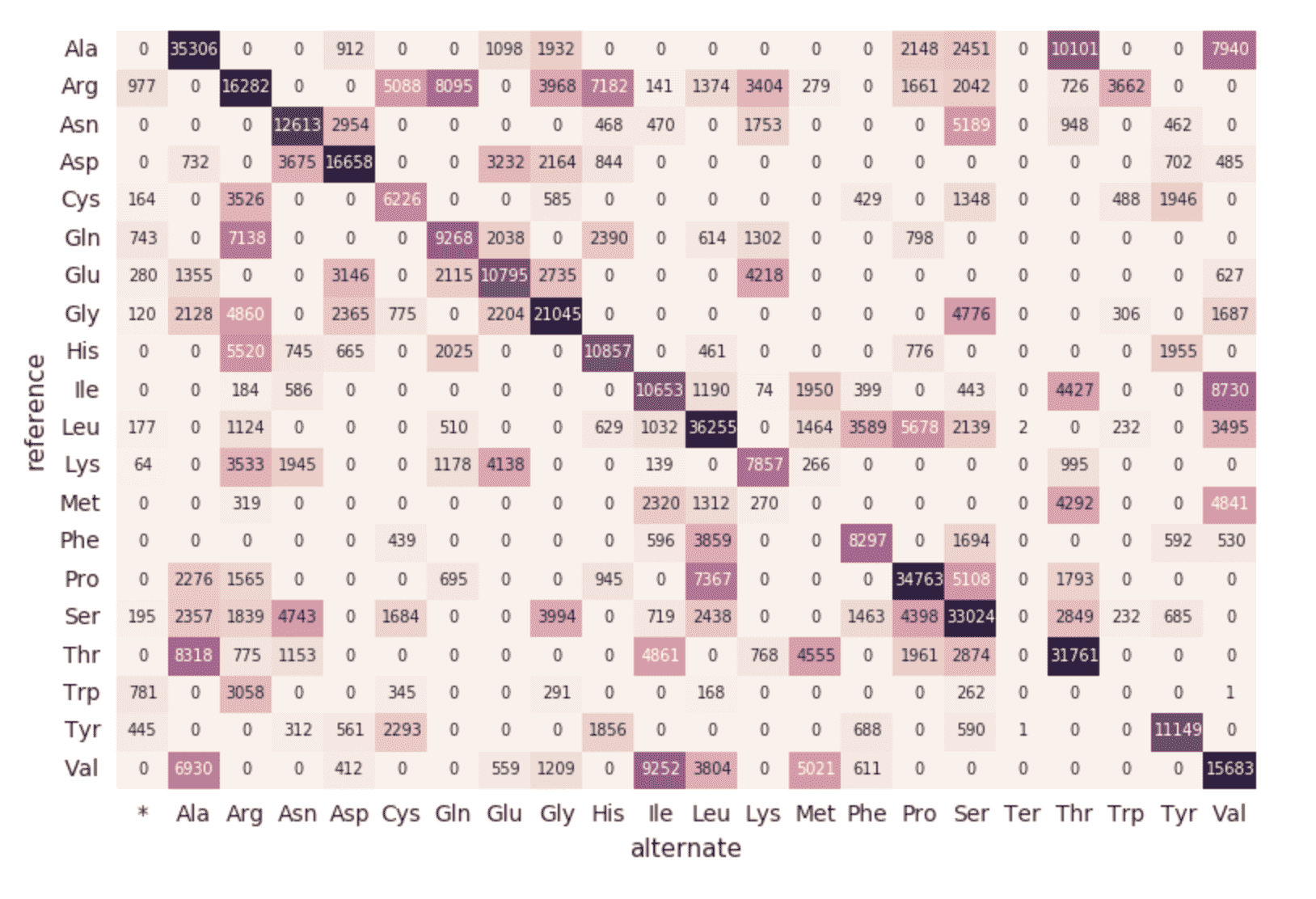
아미노산 치환 히트맵
엑솜 데이터 프레임에 이어서 다음 코드 조각을 사용하여 아미노산 치환 개수를 계산해 보겠습니다. 이전 DataFrames와 마찬가지로 또 다른 동적 DataFrame(aa_counts)을 생성하여 엑솜 데이터 프레임에서 새로운 데이터가 처리되면 이후 아미노 acid 치환 수에도 반영되도록 할 것입니다. 또한 데이터를 메모리에 쓰고 있습니다(즉, .format("memory")) 를 사용하여 60초마다 배치를 처리합니다(예 trigger(processingTime='60초'))를 설정하여 다운스트림 판다스 히트맵 코드가 히트맵을 처리하고 시각화할 수 있도록 합니다.
다음 코드 스니펫은 앞의 amino_acid_substitutions Spark 테이블을 읽고, 최대 개수를 결정하고, Spark 테이블에서 새로운 Pandas 피벗 테이블을 만든 다음 히트맵을 플로팅합니다.
연속 파이프라인으로 마이그레이션
여기까지 앞선 코드 스니펫과 시각화는 단일 sampleId에 대한 단일 실행을 나타냅니다. 하지만 Structured Streaming과 Databricks Delta를 사용하고 있으므로 이 코드를 (변경 없이) 사용하여 샘플이 파이프라인을 통과할 때 지속적으로 품질 관리 통계를 계산하는 프로덕션 데이터 파이프라인을 구축할 수 있습니다. 이를 보여주기 위해 전체 데이터 세트를 로드하는 다음 코드 스니펫을 실행할 수 있습니다.
앞의 코드 스니펫에서 설명한 것처럼 엑솜 DataFrame의 소스는 delta_stream_output 폴더에 로드된 파일입니다. 처음에는 단일 샘플아이디에 대한 파일 집합을 로드했습니다(예: 샘플아이디 = "SRS000030_SRR709972"). 앞의 코드 스니펫은 이제 생성된 모든 parquet 샘플(예 Parquet) 해당 파일들을 sampleId 별로 동일한 delta_stream_output 폴더에 증분식으로 로드합니다. 다음 애니메이션 GIF는 앞의 코드 스니펫의 축약된 출력을 보여줍니다.
https://www.youtube.com/watch?v=JPngSC5Md-Q
유전체학 파이프라인 시각화
노트북의 맨 위로 다시 스크롤하면 exomes DataFrame이 이제 새 sampleIds를 자동으로 로드하는 것을 확인할 수 있습니다. 유전체학 파이프라인의 Structured Streaming 구성 요소는 계속 실행되므로 새 파일이 delta_stream_outputpath 폴더에 로드되는 즉시 데이터를 처리합니다. Databricks Delta 형식을 사용하여 exomes DataFrame으로 스트리밍되는 데이터의 트랜잭션 일관성을 보장할 수 있습니다.
https://www.youtube.com/watch?v=Q7KdPsc5mbY
exomes DataFrame의 초기 생성과 달리 Structured Streaming 모니터링 대시보드가 이제 데이터를 로드하고 있음을 확인하세요(예: 변동하는 '입력 대 처리 속도', 변동하는 '배치 기간', '집계 상태'에서 고유 키 증가). exomes DataFrame이 처리됨에 따라 sampleIds 의 새 행(및 변이 수)을 확인하세요. 이와 동일한 작업은 연관된 돌연변이 유형별 그룹화 쿼리에서도 볼 수 있습니다.
https://www.youtube.com/watch?v=sT179SCknGM
Databricks Delta를 사용하면 유전체학 파이프라인의 모든 단계에서 새로운 데이터의 트랜잭션 일관성이 유지됩니다. 이는 파이프라인의 일관성(데이터의 일관성을 유지, 즉 모든 데이터가 “올바름”을 보장), 신뢰성(트랜잭션이 완전히 성공하거나 실패함), 실시간 업데이트 처리 기능(여러 트랜잭션을 동시에 처리할 수 있고 중단이나 장애가 데이터에 영향을 미치지 않음)을 보장하기 때문에 중요합니다. 따라서 여러 추가 ETL 단계를 거친 다운스트림 아미노산 치환 맵의 데이터도 원활하게 새로고침됩니다.
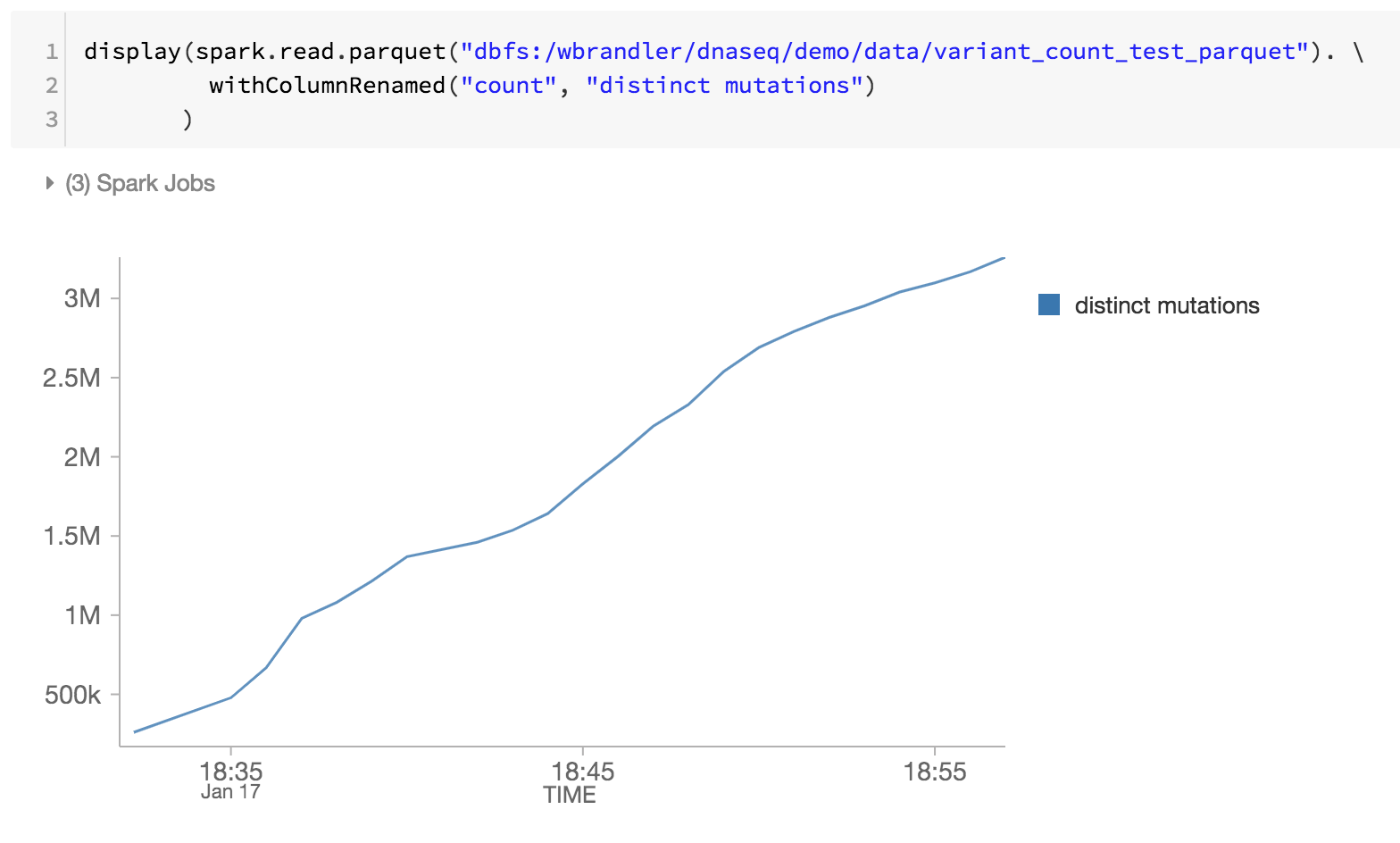
유전체학 파이프라인의 마지막 단계로, DBFS 내의 Databricks Delta parquet 파일을 검토하여 고유한 돌연변이도 모니터링하고 있습니다(예: 시간 경과에 따른 고유한 돌연변이 증가).
요약
생물정보학자 및 연구자는 Databricks 통합 분석 플랫폼을 기반으로, 특히 Databricks Delta에 중점을 두고 Databricks 유전체학 통합 분석 플랫폼을 사용하여 트랜잭션 일관성을 갖춘 분산 분석을 적용할 수 있습니다. 이러한 추상화를 통해 데이터 실무자는 유전체학 파이프라인을 단순화할 수 있습니다. 여기서는 수동 개입 없이 새로운 샘플이 처리될 때 데이터를 지속적으로 처리하는 게놈 샘플 품질 관리 파이프라인을 만들었습니다. ETL을 수행하든 정교한 분석을 수행하든, 데이터는 유전체학 파이프라인을 통해 빠르고 타격 없이 흐를 것입니다. 오늘 Databricks Delta를 사용하여 대규모 유전체학 파이프라인 단순화 노트북을 다운로드하여 직접 사용해 보세요.
대규모 유전체학 분석 시작하기:
- Unified Analytics for 유전체학 솔루션 가이드를 읽어보세요
- Databricks Delta로 대규모 유전체학 파이프라인 간소화 노트북을 다운로드하세요.
- Databricks 유전체학용 Unified Analytics 무료 체험 에 등록하세요.
감사의 말
기여해 주신 Yongsheng Huang님과 Michael Ortega님께 감사드립니다.
Delta Lake 온라인 허브를 방문하여 자세히 알아보고, 최신 코드를 다운로드하고, Delta Lake 커뮤니티에 참여하세요.