Simplifier les pipelines de génomique à grande échelle avec Databricks Delta

Obtenez un aperçu en avant-première du nouvel ebook d'O'Reilly pour le guide pas à pas dont vous avez besoin pour commencer à utiliser Delta Lake.
Essayer ce Notebook dans Databricks
Ce blog est le premier de notre série « Analyse génomique à grande échelle ». Dans cette série, nous allons montrer comment la plateforme d'analyse unifiée pour la génomique de Databricks permet aux clients d'analyser des données génomiques à l'échelle de la population. À partir du résultat de notre pipeline génomique, cette série fournira un tutoriel sur l'utilisation de Databricks pour exécuter le contrôle qualité des échantillons, le génotypage conjoint, le contrôle qualité de la cohorte et des analyses statistiques génétiques avancées.
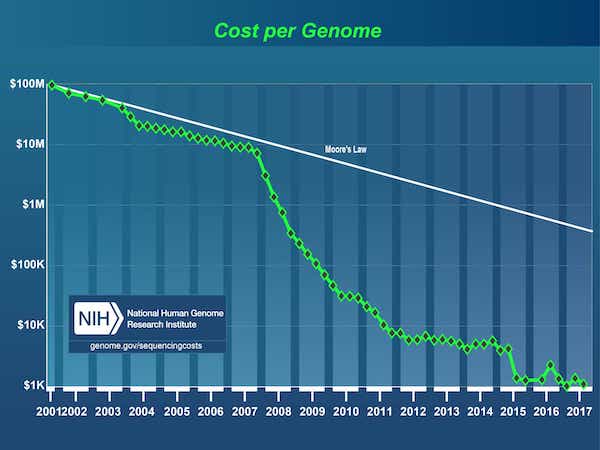
Depuis l'achèvement du Projet Génome Humain en 2003, il y a eu une explosion des données alimentée par une baisse spectaculaire du coût du séquençage de l'ADN, passant de 3 milliards de dollars1 pour le premier génome à moins de 1 000 $ aujourd'hui.
[1] Le Projet Génome Humain était un projet de 3 milliards de dollars mené par le Département de l'Énergie et les National Institutes of Health, qui a débuté en 1990 et s'est achevé en 2003.
Source : Coûts du séquençage de l'ADN : données
Par conséquent, le domaine de la génomique a atteint un stade de maturité où les entreprises ont commencé à effectuer le séquençage de l'ADN à l'échelle de la population. Cependant, le séquençage du code ADN n'est que la première étape ; les données brutes doivent ensuite être transformées dans un format adapté à l'analyse. Généralement, cette opération consiste à assembler une série d'outils de bio-informatique avec des scripts personnalisés et à traiter les données sur un nœud unique, un échantillon à la fois, jusqu'à l'obtention d'une collection de variants génomiques. Les scientifiques en bio-informatique consacrent aujourd'hui la majeure partie de leur temps à la création et à la maintenance de ces pipelines. Alors que les jeux de données génomiques ont atteint l'échelle du pétaoctet, il est devenu difficile de répondre rapidement, même aux questions simples suivantes :
- Combien d'échantillons avons-nous séquencés ce mois-ci ?
- Quel est le nombre total de variantes uniques détectées ?
- Combien de variants avons-nous observés dans les différentes classes de variation ?
Pour aggraver encore ce problème, les données de milliers d'individus ne peuvent être ni stockées, ni suivies, ni versionnées, tout en restant accessibles et requêtables. Par conséquent, les chercheurs dupliquent souvent des sous-ensembles de leurs données génomiques lorsqu'ils effectuent leurs analyses, ce qui entraîne une augmentation de l'empreinte de stockage globale et des coûts. Pour tenter de pallier ce problème, les chercheurs emploient aujourd'hui une stratégie de « gel des données », généralement sur des périodes de six mois à deux ans, pendant lesquelles ils interrompent le traitement de nouvelles données pour se concentrer sur une copie gelée des données existantes. Il n'existe aucune solution pour construire des analyses de manière incrémentale sur des périodes plus courtes, ce qui ralentit la progression de la recherche.
Il existe un besoin impérieux d'outils robustes capables d'ingérer des données génomiques à l'échelle industrielle, tout en offrant aux scientifiques la flexibilité nécessaire pour explorer les données, itérer sur leurs pipelines analytiques et en tirer de nouveaux insights.
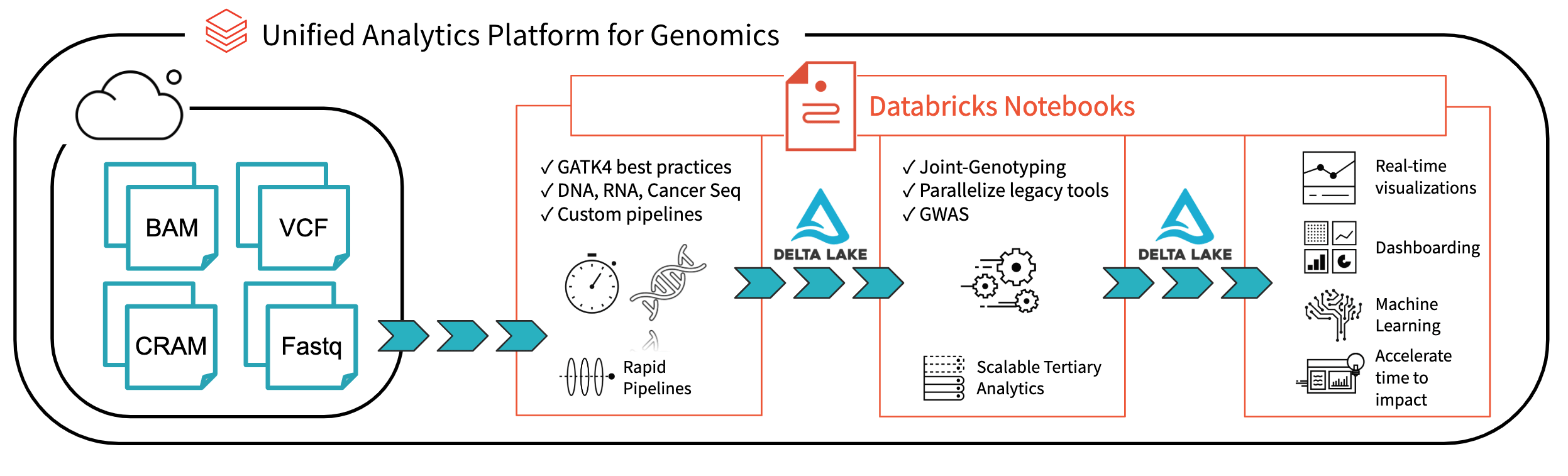
Fig 1. Architecture pour l'analyse génomique de bout en bout avec Databricks
Avec Databricks Delta : Un système de gestion unifié pour l'Analytique Big Data en temps réel, la plateforme Databricks a fait un grand pas en avant pour résoudre les problèmes de gouvernance des données, d'accès aux données et d'analyse des données auxquels les chercheurs sont aujourd'hui confrontés. Avec Databricks Delta Lake, vous pouvez stocker toutes vos données génomiques en un seul endroit et créer des analyses qui se mettent à jour en temps réel à mesure que de nouvelles données sont ingérées. Associé aux optimisations de notre plateforme d'analyse unifiée pour la génomique (UAP4G) pour la lecture, l'écriture et le traitement des formats de fichiers génomiques, nous proposons une solution de bout en bout pour les workflows de pipelines génomiques. L'architecture UAP4G offre de la flexibilité, permettant aux clients d'intégrer leurs propres pipelines et de développer leur propre analytique tertiaire. À titre d'exemple, nous avons mis en évidence le tableau de bord suivant qui présente des métriques et des visualisations de contrôle qualité pouvant être calculées et présentées de manière automatisée et personnalisées pour répondre à vos besoins spécifiques.
https://www.youtube.com/watch?v=73fMhDKXykU
Dans la suite de ce blog, nous décrirons les étapes que nous avons suivies pour créer le tableau de bord de contrôle qualité ci-dessus, qui se met à jour en temps réel à mesure que le traitement des échantillons se termine. En utilisant un pipeline basé sur Delta pour le traitement des données génomiques, nos clients peuvent désormais exploiter leurs pipelines en bénéficiant d'une visibilité en temps réel, échantillon par échantillon. Avec les Notebooks Databricks (et des intégrations telles que GitHub et MLflow), ils peuvent suivre et versionner les analyses de manière à garantir la reproductibilité de leurs résultats. Leurs bio-informaticiens peuvent consacrer moins de temps à la maintenance des pipelines et passer plus de temps à faire des découvertes. Nous considérons l'UAP4G comme le moteur qui pilotera la transformation des analyses ad hoc vers la génomique de production à l'échelle industrielle, permettant ainsi de mieux comprendre le link entre la génétique et la maladie.
Lire les données d'échantillon
Commençons par lire les données de variation d'une petite cohorte d'échantillons ; l'instruction suivante lit les données pour un sampleId spécifique et les enregistre au format Databricks Delta (dans le dossier delta_stream_output).
Remarque : le dossier annotations_etl_parquet contient des annotations générées à partir de l'ensemble de données 1000 génomes et stockées au format Parquet. L'ETL et le traitement de ces annotations ont été effectués à l'aide de la plateforme d'analyse unifiée pour la génomique de Databricks.
Start le streaming de la table Databricks Delta
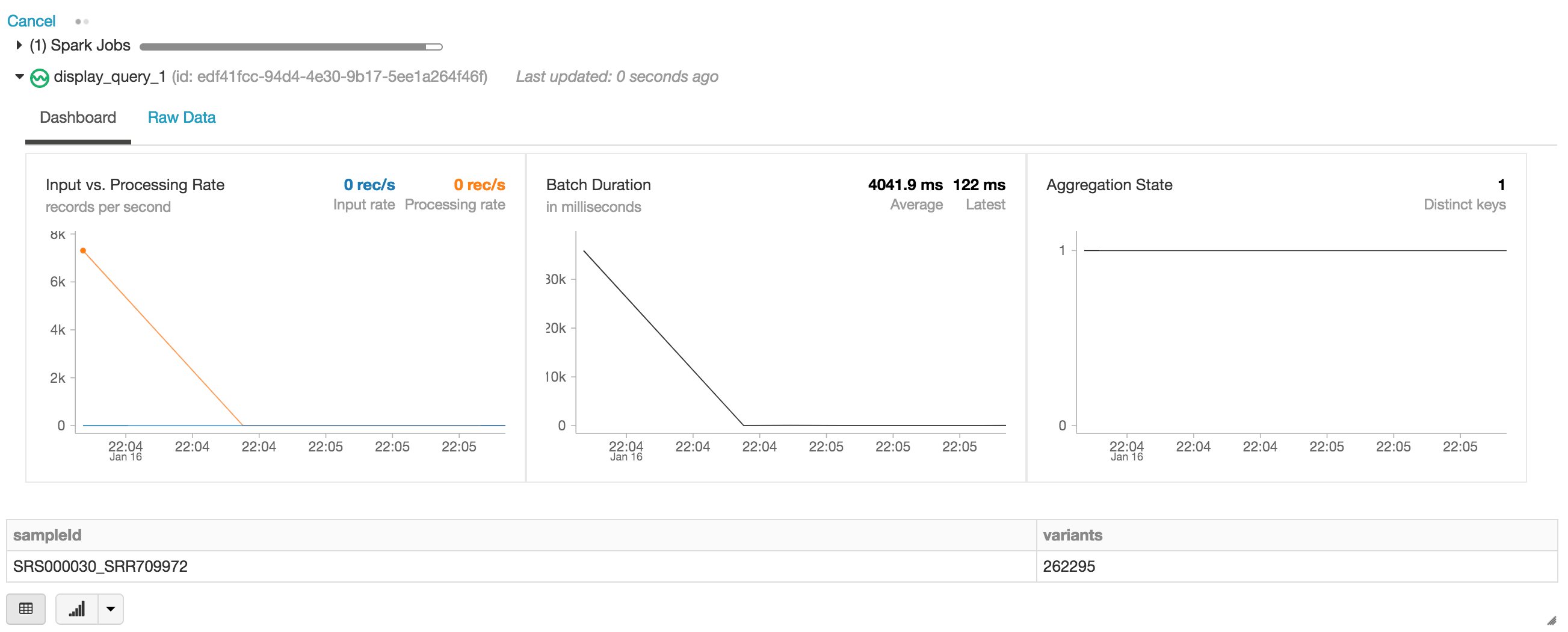
Dans l'instruction suivante, nous créons le DataFrame Apache Spark des exomes, qui lit un stream de données (via readStream) au format Databricks Delta. Il s'agit d'un DataFrame en exécution continue ou dynamique, c'est-à-dire que le DataFrame exomes chargera de nouvelles données au fur et à mesure de leur écriture dans le dossier delta_stream_output. Pour afficher le DataFrame exomes, nous pouvons exécuter une query de DataFrame pour trouver le nombre de variants regroupés par le sampleId.
Lors de l'exécution de l'instruction display, le Notebook Databricks fournit un tableau de bord de streaming pour surveiller les tâches de streaming. Les résultats de l'instruction display (c'est-à-dire le nombre de variants par sample_id) se trouvent juste en dessous du job de streaming.
Continuons de répondre à notre série de questions initiale en exécutant d'autres queries DataFrame basées sur notre DataFrame exomes.
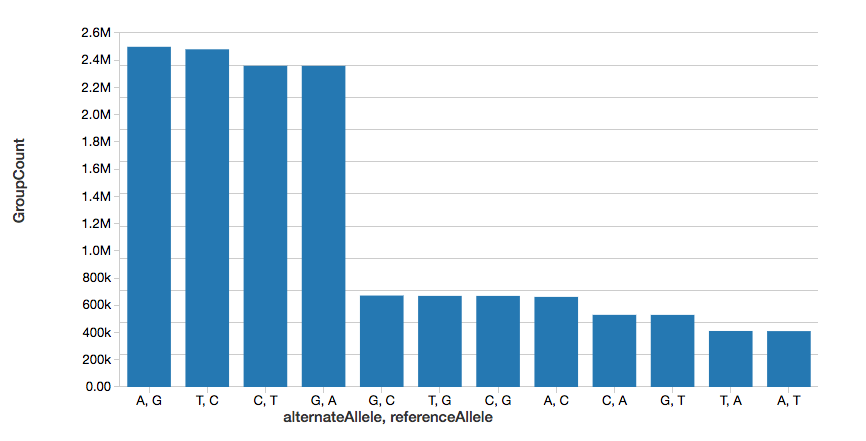
Nombre de variants mononucléotidiques
Pour poursuivre l'exemple, nous pouvons rapidement calculer le nombre de variants nucléotidiques uniques (SNV), comme l'illustre le Graphe suivant.
Notez que la commande display fait partie du workspace Databricks et vous permet de visualiser votre DataFrame à l'aide des visualisations Databricks (c'est-à-dire aucun codage requis).
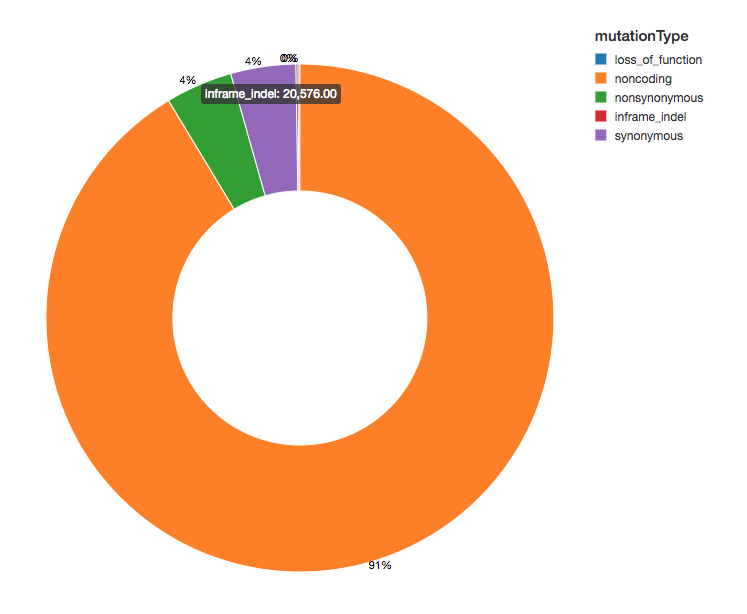
Nombre de variantes
Puisque nous avons annoté nos variants avec des effets fonctionnels, nous pouvons poursuivre notre analyse en examinant la répartition des effets des variants que nous observons. La majorité des variants détectés flanquent des régions qui codent pour des protéines ; il s'agit de variants non codants.
Démarrer avec l'ETL

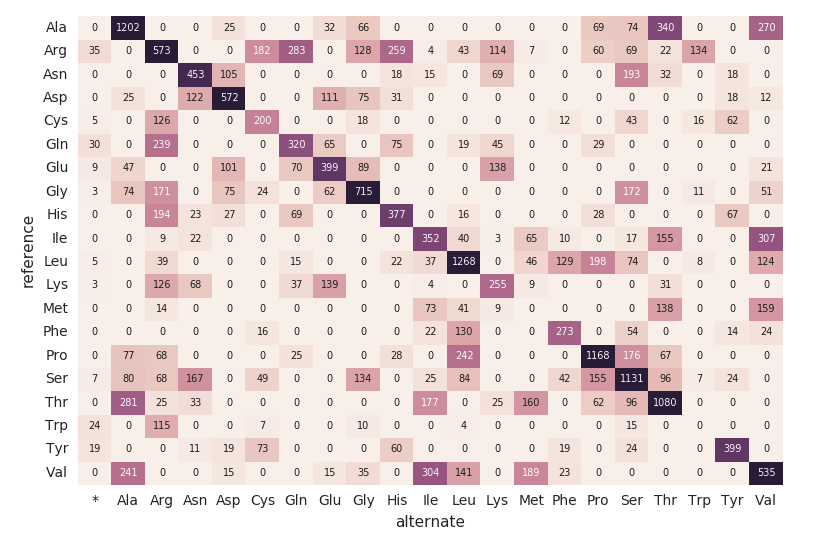
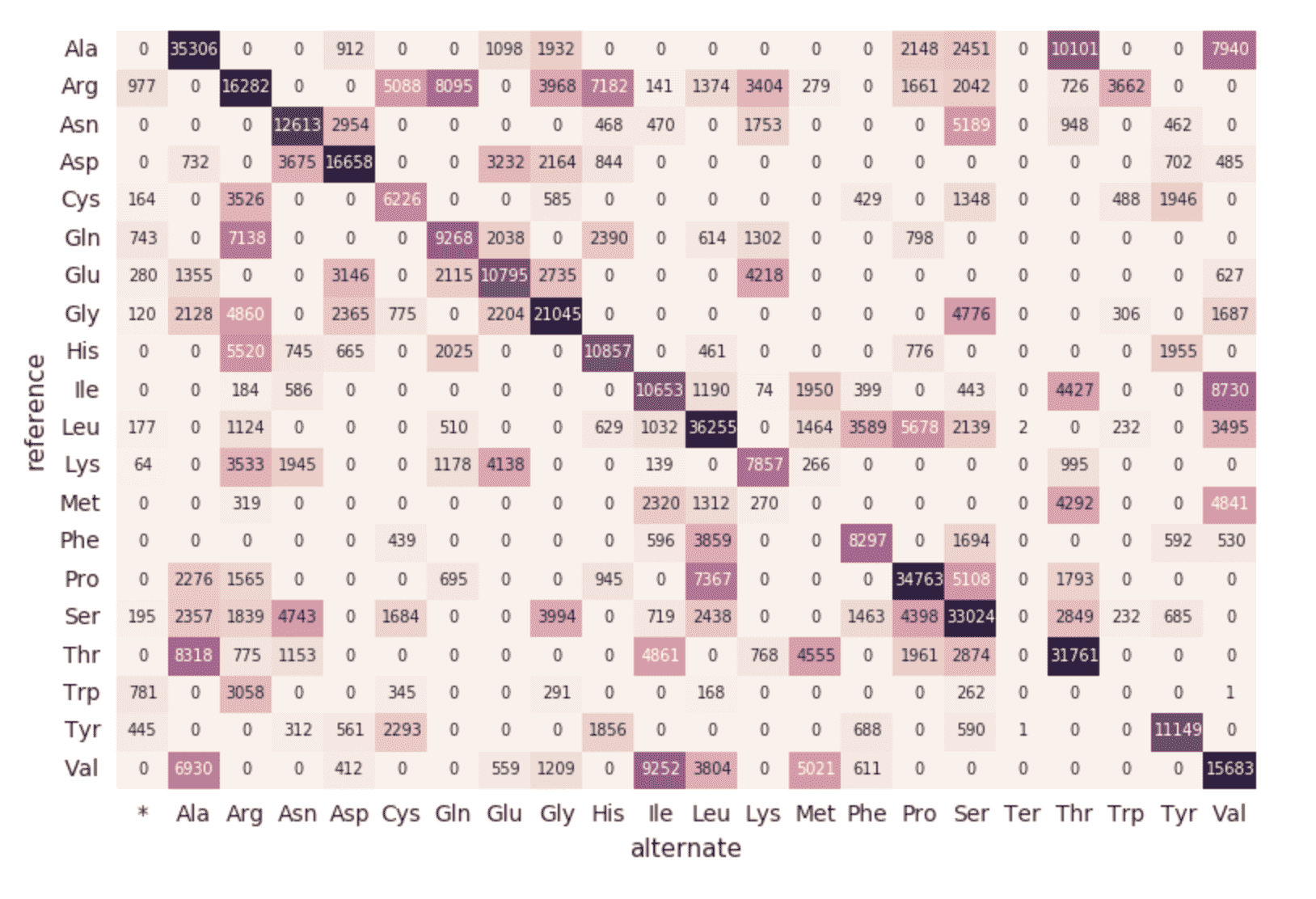
Carte thermique de substitution d'acides aminés
Poursuivons avec notre DataFrame exomes et calculons les décomptes de substitutions d'aminoacides à l'aide du fragment de code suivant. Comme pour les DataFrames précédents, nous allons créer un autre DataFrame dynamique (aa_counts) de sorte que lorsque de nouvelles données sont traitées par le DataFrame exomes, elles soient également répercutées dans les décomptes de substitutions d'acides aminés. Nous écrivons également les données en mémoire (c'est-à-dire .format("memory")) et traitons les batches toutes les 60 secondes (c'est-à-dire trigger(processingTime=’60 seconds’)) afin que le code de la heatmap Pandas en aval puisse traiter et visualiser la heatmap.
L'extrait de code suivant lit la table Spark amino_acid_substitutions précédente, détermine le nombre maximal, crée un nouveau tableau croisé dynamique Pandas à partir de la table Spark, puis trace la carte thermique.
Migration vers un pipeline continu
Jusqu'à présent, les extraits de code et les visualisations précédents représentent une seule exécution pour un seul sampleId. Mais comme nous utilisons Structured Streaming et Databricks Delta, ce code peut être utilisé (sans aucune modification) pour construire un pipeline de données de production qui calcule des statistiques de contrôle qualité en continu au fur et à mesure que les échantillons transitent par notre pipeline. Pour le démontrer, nous pouvons exécuter l'extrait de code suivant qui chargera l'ensemble de notre dataset.
Comme décrit dans les extraits de code précédents, la source du DataFrame exomes correspond aux fichiers chargés dans le dossier delta_stream_output. Initialement, nous avions chargé un ensemble de fichiers pour un seul sampleId (c'est-à-dire, sampleId = “SRS000030_SRR709972”). L'extrait de code précédent prend désormais tous les échantillons Parquet générés (c'est-à-dire parquets) et charge incrémentalement ces fichiers par sampleId dans le même dossier delta_stream_output. Le GIF animé suivant montre la sortie abrégée de l'extrait de code précédent.
https://www.youtube.com/watch?v=JPngSC5Md-Q
Visualisation de votre pipeline génomique
Lorsque vous revenez en haut de votre notebook, vous remarquerez que le DataFrame exomes charge désormais automatiquement les nouveaux sampleIds. Comme le composant de streaming structuré de notre pipeline de génomique s'exécute en continu, il traite les données dès que de nouveaux fichiers sont chargés dans le dossier delta_stream_outputpath. En utilisant le format Databricks Delta, nous pouvons garantir la cohérence transactionnelle des données de streaming dans le DataFrame exomes.
https://www.youtube.com/watch?v=Q7KdPsc5mbY
Contrairement à la création initiale de notre DataFrame exomes, remarquez comment le tableau de bord de monitoring du streaming structuré charge maintenant des données (c.-à-d., la fluctuation du « taux d'entrée/traitement », la fluctuation de la « durée de batch » et l'augmentation des clés distinctes dans l'« état des agrégations »). Pendant le traitement du DataFrame exomes, vous remarquerez l'apparition de nouvelles lignes de sampleIds (et le nombre de variants). Cette même action peut également être observée pour la associée group by mutation type query.
https://www.youtube.com/watch?v=sT179SCknGM
Avec Databricks Delta, toute nouvelle donnée est cohérente sur le plan transactionnel à chaque étape de notre pipeline de génomique. Ceci est important, car cela garantit que votre pipeline est cohérent (maintient la cohérence de vos données, c'est-à-dire garantit que toutes les données sont « correctes »), fiable (la transaction réussit ou échoue complètement) et peut gérer les mises à jour en temps réel (la capacité de gérer de nombreuses transactions simultanément et toute panne ou défaillance n'aura pas d'impact sur les données). Ainsi, même les données de notre carte de substitution d'acides aminés en aval (qui ont subi un certain nombre d'étapes ETL supplémentaires) sont actualisées de manière transparente.
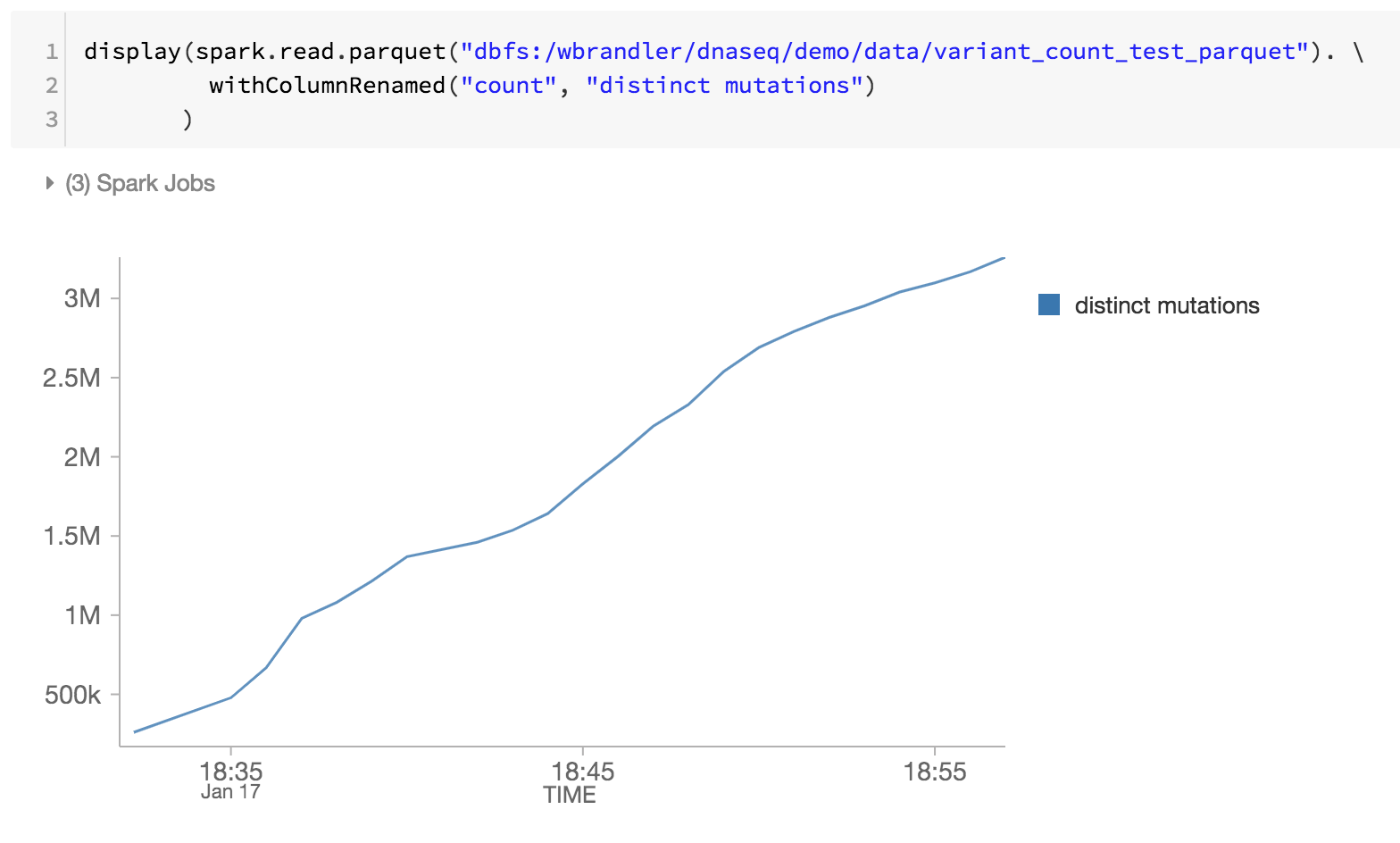
En tant que dernière étape de notre pipeline de génomique, nous effectuons le monitoring des mutations distinctes en examinant les fichiers parquet Databricks Delta dans DBFS (c'est-à-dire l'augmentation des mutations distinctes au fil du temps).
Résumé
En s'appuyant sur les fondations de la Databricks plateforme d'analyse unifiée - et en particulier sur Databricks Delta - les bio-informaticiens et les chercheurs peuvent appliquer des analytiques distribuées avec une cohérence transactionnelle à l'aide de Databricks plateforme d'analyse unifiée pour la génomique. Ces abstractions permettent aux praticiens des données de simplifier les pipelines génomiques. Ici, nous avons créé un pipeline de contrôle qualité des échantillons génomiques qui traite les données en continu au fur et à mesure que de nouveaux échantillons sont traités, sans intervention manuelle. Que vous effectuiez des opérations d'ETL ou des analyses sophistiquées, vos données circuleront dans votre pipeline génomique rapidement et sans disruption. Essayez-le vous-même dès aujourd'hui en téléchargeant le notebook Simplification des pipelines génomiques à grande échelle avec Databricks Delta.
start l'analyse de la génomique à Monter en charge :
- Consultez notre guide de la solutionsur l'analyse unifiée pour la génomique
- Téléchargez le Notebook : Simplification des pipelines génomiques à l'échelle avec Databricks Delta
- Inscrivez-vous pour un essai gratuit de Databricks Unified Analytics Platform pour la génomique
Remerciements
Merci à Yongsheng Huang et Michael Ortega pour leurs contributions.
Visitez le hub en ligne de Delta Lake pour en savoir plus, télécharger le dernier code et rejoindre la communauté Delta Lake.
Ne manquez jamais un article Databricks
Sign up
Et ensuite ?

Data Science e ML
31 octobre 2023/9 min de lecture