Accelerare la scoperta di farmaci: dai file FASTA alle informazioni dettagliate della GenAI su Databricks
Come creare una pipeline end-to-end che combina ingegneria dei dati, Protein Language Models e GenAI sulla Databricks Platform.
Summary
- Elabora dati biologici su larga scala utilizzando le pipeline dichiarative di Lakeflow per trasformare le sequenze proteiche FASTA grezze in tabelle pronte per l'analisi in Unity Catalog.
- Classifica le proteine con modelli transformer sfruttando ProtBERT, un modello linguistico per le proteine, per identificare le proteine di trasporto della membrana, bersagli farmacologici chiave.
- Esegui query sugli approfondimenti sulle proteine in linguaggio naturale tramite le AI Functions che collegano gli LLM direttamente ai tuoi dati, consentendo ai ricercatori di esplorare promettenti farmaci candidati in modo conversazionale.
Lo sviluppo di farmaci è notoriamente lento e costoso. Il ciclo di vita medio di Ricerca e Sviluppo (R&D) dura 10-15 anni e una parte significativa dei candidati non supera le sperimentazioni cliniche. Un importante collo di bottiglia è stato l'identificazione dei giusti bersagli proteici nella fase iniziale del processo.
Le proteine sono le "molecole operative" degli organismi viventi: catalizzano le reazioni, trasportano molecole e fungono da bersaglio per la maggior parte dei farmaci moderni. La capacità di classificare rapidamente le proteine, comprenderne le proprietà e identificare candidati poco studiati potrebbe accelerare notevolmente il processo di scoperta (ad es. Wozniak et al., 2024, Nature Chemical Biology).
È qui che la convergenza tra ingegneria dei dati, machine learning (ML) e IA generativa diventa trasformativa. Di fatto, è possibile creare questa intera pipeline su un'unica piattaforma: la Databricks Data Intelligence Platform.
Cosa stiamo costruendo
Il nostro AI-Driven Drug Discovery Solution Accelerator dimostra un flusso di lavoro end-to-end attraverso quattro processi chiave:
- Ingestione ed elaborazione dei dati: oltre 500.000 sequenze proteiche vengono acquisite ed elaborate da UniProt.
- Classificazione basata su AI: viene utilizzato un modello transformer per classificare queste proteine come idrosolubili o di trasporto di membrana.
- Generazione di approfondimento: i dati proteici vengono arricchiti con approfondimenti di ricerca generati da LLM.
- Esplorazione in linguaggio naturale: tutti i dati elaborati e arricchiti i dati sono resi accessibili tramite una dashboard e un ambiente basati sull'IA che supportano l'interrogazione in linguaggio naturale.
Analizziamo ogni fase:
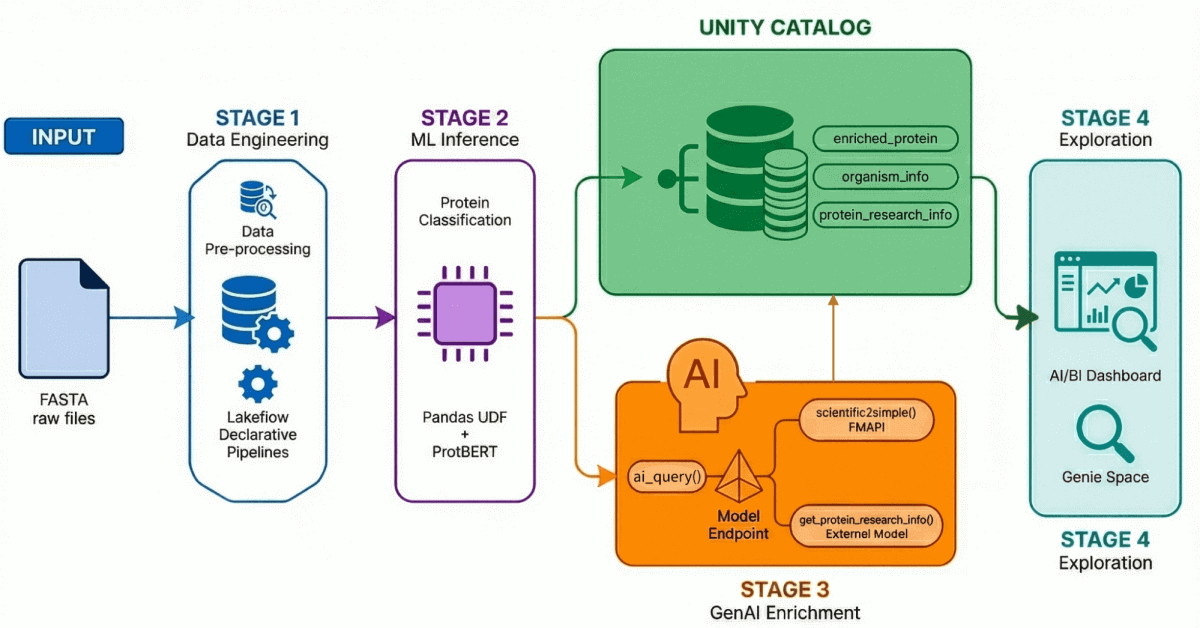
Fase 1: Data ingegneria con Lakeflow Declarative Pipelines
I dati biologici grezzi raramente arrivano in un formato pulito e pronto per l'analisi. I nostri dati di origine si presentano sotto forma di file FASTA, un formato standard per la rappresentazione delle sequenze proteiche, simile al seguente:
Per un occhio inesperto, questi dati di sequenza sono quasi impossibili da interpretare: una fitta stringa di codici di amminoacidi a lettera singola. Tuttavia, al termine di questa pipeline, i ricercatori possono effettuare query su questi stessi dati in linguaggio naturale, ponendo domande come "Mostrami le proteine di membrana poco studiate nell'uomo con un'elevata confidenza di classificazione" e ricevendo in cambio informazioni dettagliate.
Utilizzando Lakeflow Declarative Pipelines, creiamo un'architettura medallion che affina progressivamente questi dati:
- Livello Bronzo: Ingestione grezza di file FASTA utilizzando BioPython, estraendo ID e sequenze.
- Livello Silver: parsing e strutturazione: estraiamo nomi di proteine, informazioni sull'organismo, nomi di geni e altri metadati usando trasformazioni regex.
- Livello Gold/arricchito: dati curati e pronti per l'analisi, arricchiti con metriche derivate come il peso molecolare e pronti per dashboard, modelli di ML e ricerca a valle. Questo è il livello affidabile che analisti e scienziati interrogano direttamente.
Il risultato: dati sulle proteine puliti e governati in Unity Catalog, pronti per le analitiche e l'ML downstream. Fondamentalmente, la provenienza dei dati che si estende oltre questa fase fino alle altre (evidenziate di seguito) fornisce un valore incredibile per la riproducibilità scientifica.
Fase 2: Classificazione delle proteine con modelli Transformer
Non tutte le proteine sono uguali nel campo della scoperta di farmaci. Le proteine di trasporto della membrana, quelle integrate nelle membrane cellulari, sono bersagli farmacologici particolarmente importanti perché controllano l'ingresso e l'uscita dalle cellule.
Utilizziamo ProtBERT-BFD, un modello linguistico proteico basato su BERT del Rostlab, ottimizzato in modo specifico per la classificazione delle proteine di membrana. Questo modello tratta le sequenze di amminoacidi come un linguaggio, apprendendo le relazioni contestuali tra i residui per prevedere la funzione delle proteine.
Il modello restituisce una classificazione (come Membrana o Solubile) insieme a un punteggio di confidenza, che viene riscritto in Unity Catalog per il filtraggio e l'analisi a valle.
Fase 3: Arricchimento dei dati con GenAI
La classificazione ci dice che cos'è una proteina. Ma i ricercatori devono sapere perché è importante, ossia quali sono le ricerche più recenti. Dove sono le lacune? Si tratta di un bersaglio farmacologico poco esplorato?
È qui che entrano in gioco gli LLM. Sfruttando sia l'API Foundational Model di Databricks sia gli endpoint dei modelli esterni, creiamo AI Functions registrate che arricchiscono i record delle proteine con il contesto della ricerca.
Fase 4: Esplorazione del linguaggio naturale
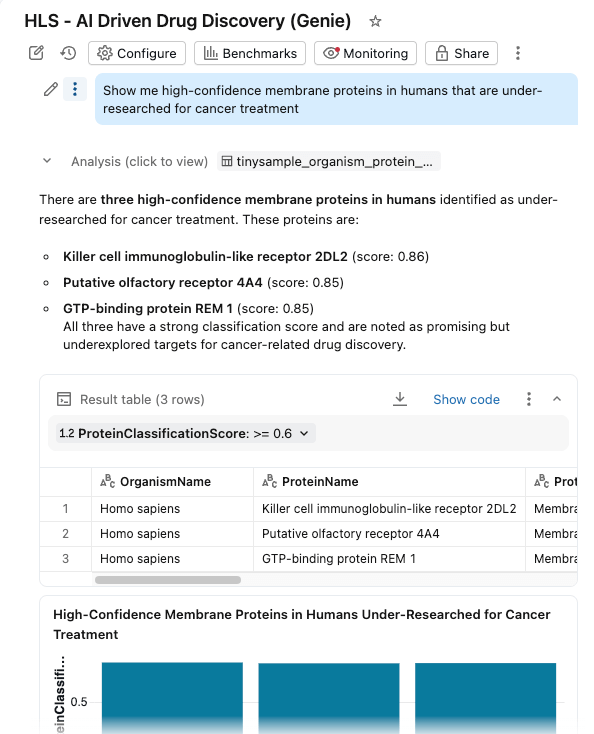
Riuniamo tutto in una dashboard di AI/BI con Genie Space abilitato.
I ricercatori ora possono:
- Filtra proteine per organismo, punteggio di classificazione e tipo di proteina
- Esplora le distribuzioni dei pesi molecolari e della confidenza della classificazione
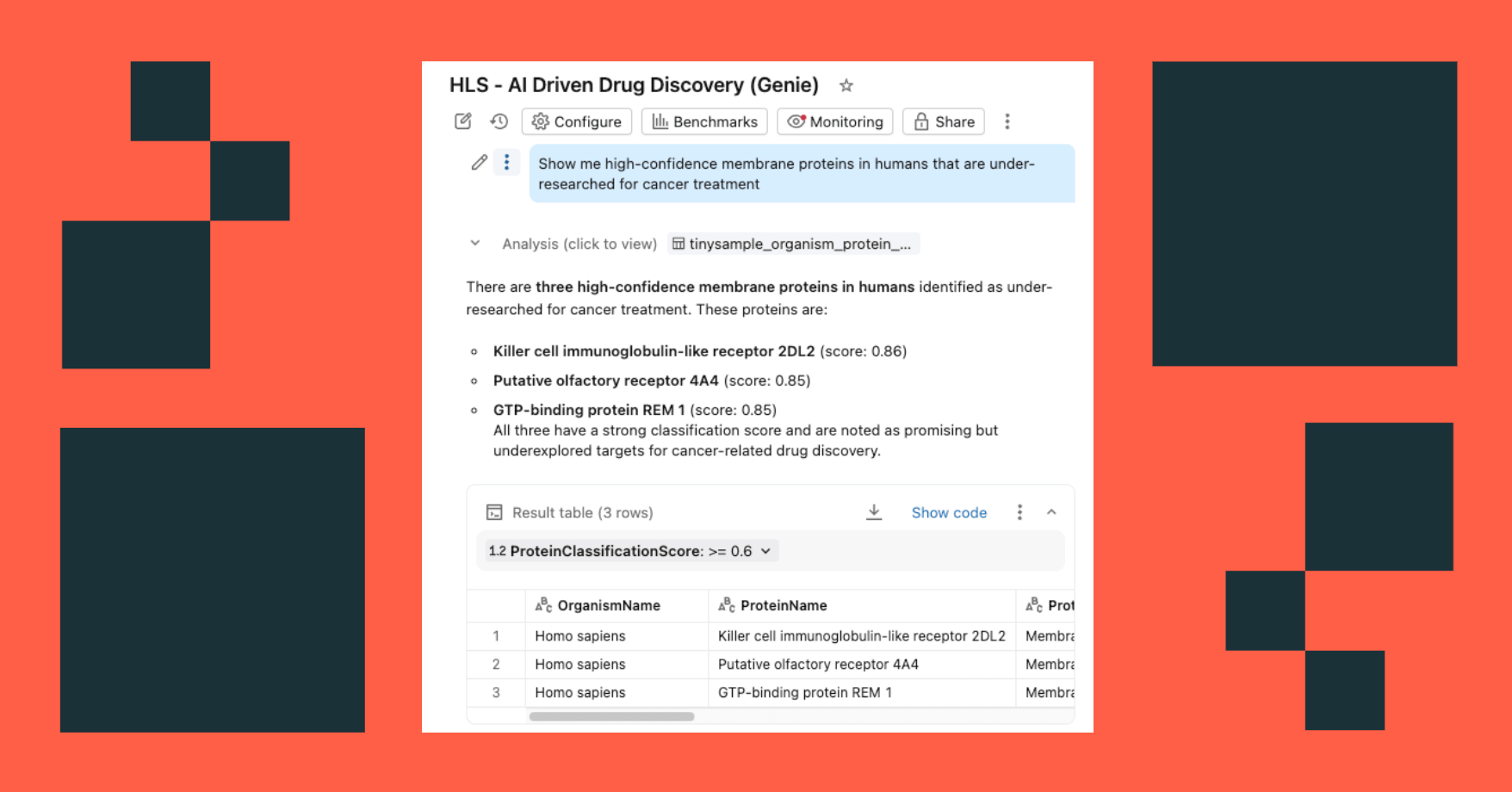
- Poni domande in linguaggio naturale: "Mostrami le proteine di membrana ad alta confidenza nell'uomo che sono poco studiate per il trattamento del cancro"
La dashboard interroga le stesse tabelle governate in Unity Catalog, con le Funzioni IA che forniscono un arricchimento on-demand (o elaborato in batch).
La data intelligence rimodella i settori industriali

La potenza di una piattaforma unificata
Ciò che rende interessante questa soluzione non è dovuto a un singolo componente, ma al fatto che tutto viene eseguito su un'unica piattaforma:
| Capacità | Funzionalità di Databricks |
|---|---|
| Ingestione dati & ETL | Lakeflow Declarative Pipelines |
| Governance dei dati | Unity Catalog |
| Inferenza ML | GPU compute |
| Integrazione LLM | FMAPI + Modelli esterni + Funzioni IA |
| Analisi | Databricks SQL |
| Esplorazione | Dashboard AI/BI + AI/BI Genie Space |
Fondamentalmente, non avviene alcun movimento di dati tra i sistemi. Nessuna infrastruttura MLOps separata. Nessuno strumento di BI disconnesso. La sequenza proteica che entra nella pipeline fluisce attraverso la trasformazione, la classificazione e l'arricchimento, fino a diventare interrogabile in linguaggio naturale, il tutto all'interno dello stesso ambiente governato.
L'acceleratore di soluzioni completo è disponibile su GitHub:
github.com/databricks-industry-solutions/ai-driven-drug-discovery
Prossimi passi
Questo acceleratore dimostra l'arte del possibile. In produzione, potresti estenderlo a:
- Elabora l'intero database UniProt con endpoint di throughput con provisioning
- Aggiungi altri modelli di classificazione (aperti o personalizzati) per diverse proprietà delle proteine
- Crea pipeline RAG sulla letteratura scientifica per ottenere risposte LLM più fondate.
- Integrazione con i flussi di lavoro di simulazione molecolare a valle
- Connettiti alla previsione della struttura proteica (AlphaFold/ESMFold) per aggiungere un contesto strutturale 3D alle proteine classificate
- Estendi ad altri formati genomici (FASTQ, VCF, BAM) utilizzando Glow per il sequenziamento su larga scala e l'analisi delle varianti
Le basi sono state gettate. La piattaforma è unificata. L'unico limite è la scienza che si vuole accelerare. Comincia oggi stesso!
(Questo post sul blog è stato tradotto utilizzando strumenti basati sull'intelligenza artificiale) Post originale
Non perdere mai un post di Databricks
Sign up
Cosa succederà adesso?

Saúde e ciências da vida
1 dicembre 2025/5 min di lettura
Databricks e NVIDIA: impulsionando a próxima geração de AI para as indústrias
Saúde e ciências da vida
21 gennaio 2026/14 min di lettura