Skalierung von SHAP-Berechnungen mit PySpark und Pandas-UDFs

Motivation
Mit der zunehmenden Verbreitung von Machine Learning (ML)- und insbesondere Deep Learning (DL)-Modellen bei der Entscheidungsfindung wird es immer wichtiger, die Blackbox zu durchschauen und wichtige Geschäftsentscheidungen auf der Grundlage der Ergebnisse solcher Modelle zu begründen. Wenn zum Beispiel ein ML-Modell den Kreditantrag eines Kunden ablehnt oder einem bestimmten Kunden ein Kreditrisiko bei der Peer-to-Peer-Kreditvergabe zuweist, könnte eine Erklärung der Gründe für diese Entscheidung ein wirksames Instrument sein, um die Anpassung der Modelle zu fördern. In vielen Fällen ist interpretierbare ML nicht nur eine Geschäftsanforderung, sondern auch eine gesetzliche Anforderung, um zu verstehen, warum eine bestimmte Entscheidung oder Option für einen Kunden getroffen wurde. SHapley Additive exPlanations (SHAP) ist ein wichtiges Werkzeug, um KI zu erklären und Vertrauen in die Ergebnisse von ML-Modellen und neuronalen Netzen bei der Lösung von Geschäftsproblemen zu schaffen.
SHAP ist ein hochmodernes Framework zur Modellerklärung, das auf der Spieltheorie basiert. Bei diesem Ansatz wird für jeden der Datenpunkte in Ihrem Dataset eine lineare Beziehung zwischen den Features eines Modells und der Modellausgabe ermittelt. Mit diesem Framework können Sie die Ausgabe Ihres Modells global oder lokal interpretieren. Globale Interpretierbarkeit hilft Ihnen zu verstehen, wie stark jedes Feature positiv oder negativ zu den Ergebnissen beiträgt. Andererseits hilft Ihnen die lokale Interpretierbarkeit, die Auswirkung jedes Features für eine bestimmte Beobachtung zu verstehen.
Die gängigsten SHAP-Implementierungen, die in der Data Science Community weit verbreitet sind, werden auf Single-Node-Maschinen ausgeführt, d. h. sie führen alle Berechnungen auf einem einzigen Kern aus, unabhängig davon, wie viele Kerne verfügbar sind. Sie nutzen daher nicht die Vorteile verteilter Berechnungen und sind durch die Einschränkungen eines einzelnen Kerns begrenzt.
In diesem Beitrag zeigen wir eine einfache Möglichkeit, SHAP-Wert-Berechnungen über mehrere Maschinen hinweg zu parallelisieren, insbesondere für die lokale Interpretierbarkeit. Anschließend erklären wir, wie diese Lösung mit der wachsenden Anzahl von Zeilen und Spalten im Dataset skaliert. Abschließend beleuchten wir einige unserer Erkenntnisse darüber, was bei der Parallelisierung von SHAP-Berechnungen mit Spark funktioniert und was man vermeiden sollte.
Single-Node-SHAP
Um Erklärbarkeit zu realisieren, wandelt SHAP ein Modell in einen Explainer um; einzelne Modellvorhersagen werden dann erklärt, indem der Explainer auf sie angewendet wird. Es gibt mehrere Implementierungen von SHAP-Wert-Berechnungen in verschiedenen Programmiersprachen, darunter eine beliebte in Python. Mit dieser Implementierung können Sie einen für Ihr Modell geeigneten Explainer anwenden, um Erklärungen für jede Beobachtung zu erhalten. Der folgende Codeausschnitt veranschaulicht, wie ein TreeExplainer auf einen Random Forest Classifier angewendet wird.
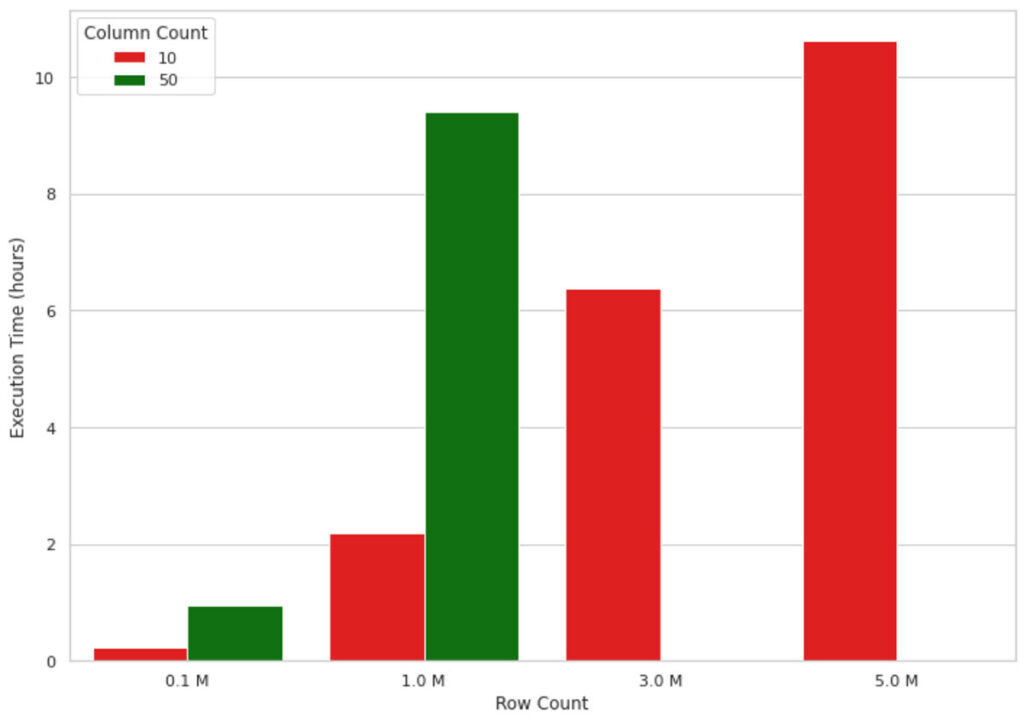
Diese Methode funktioniert gut für kleine Datenmengen, aber wenn es darum geht, die Ausgabe eines ML-Modells für Millionen von Datensätzen zu erklären, skaliert sie aufgrund der Single-Node-Natur der Implementierung nicht gut. Die Visualisierung in Abbildung 1 unten zeigt zum Beispiel den Anstieg der Ausführungszeit einer SHAP-Wert-Berechnung auf einer Single-Node-Maschine (4 Kerne und 30,5 GB Speicher) bei einer zunehmenden Anzahl von Datensätzen. Der Maschine ging bei Datenformen mit mehr als 1 Mio. Zeilen und 50 Spalten der Speicher aus, daher fehlen diese Werte in der Abbildung. Wie Sie sehen können, wächst die Ausführungszeit fast linear mit der Anzahl der Datensätze, was in realen Szenarien nicht tragbar ist. Zum Beispiel 10 Stunden zu warten, um zu verstehen, warum ein Machine-Learning-Modell eine Vorhersage getroffen hat, ist in vielen Geschäftsumgebungen weder effizient noch akzeptabel.
Eine Möglichkeit, dieses Problem zu lösen, ist die Verwendung einer approximativen Berechnung. Sie können das Argument approximate in der Methode shap_values auf True setzen. Auf diese Weise haben die unteren Splits im Baum höhere Gewichtungen, und es gibt keine Garantie, dass die SHAP-Werte mit der exakten Berechnung übereinstimmen. Dadurch werden die Berechnungen beschleunigt, aber Sie erhalten am Ende möglicherweise eine ungenaue Erklärung Ihrer Modellausgabe. Außerdem ist das approximative Argument nur in TreeExplainers verfügbar.
Ein alternativer Ansatz wäre, ein verteiltes Verarbeitungssystem wie Apache Spark™ zu nutzen, um die Anwendung des Explainer auf mehrere Kerne zu parallelisieren.
Skalierung von SHAP-Berechnungen mit PySpark
Um SHAP-Berechnungen zu verteilen, arbeiten wir mit dieser Python-Implementierung und Pandas UDFs in PySpark. Wir verwenden das kddcup99 Dataset, um einen Netzwerk-Intrusion-Detektor zu erstellen, ein prädiktives Modell, das zwischen schlechten Verbindungen, sogenannten Intrusionen oder Angriffen, und guten, normalen Verbindungen unterscheiden kann. Dieses Dataset ist bekanntermaßen fehlerhaft für Zwecke der Intrusion Detection. In diesem Beitrag konzentrieren wir uns jedoch rein auf die SHAP-Wert-Berechnungen und nicht auf die Semantik des zugrunde liegenden ML-Modells.
Bei den beiden Modellen, die wir für unsere Experimente entwickelt haben, handelt es sich um einfache Random-Forest-Klassifikatoren, die auf Datensätzen mit 10 und 50 Features trainiert wurden, um die Skalierbarkeit der Lösung bei unterschiedlichen Spaltengrößen zu zeigen. Bitte beachte, dass das Original-Dataset weniger als 50 Spalten hat und wir einige dieser Spalten repliziert haben, um unser gewünschtes Datenvolumen zu erreichen. Die Datenmengen, mit denen wir experimentiert haben, reichen von 4 MB bis 1,85 GB.
Bevor wir in den Code eintauchen, wollen wir einen kurzen Überblick darüber geben, wie Spark Dataframes und UDFs funktionieren. Spark-Dataframes werden (nach Zeilen) über einen Cluster verteilt. Jede Gruppierung von Zeilen wird als Partition bezeichnet und jede Partition kann (by default) von einem Kern bearbeitet werden. Auf diese Weise erreicht Spark im Grunde eine parallele Verarbeitung. Pandas UDFs sind eine natürliche Wahl, da Pandas leicht in SHAP eingespeist werden kann und performant ist. Eine Pandas-UDF, die auch als vektorisierte UDF bezeichnet wird, bietet uns eine bessere Leistung als Python-UDFs, da sie Apache Arrow zur Optimierung der Datenübertragung nutzt.
Der folgende Codeschnipsel zeigt, wie du die Anwendung eines Explainer mit einer Pandas UDF in PySpark parallelisieren kannst. Wir definieren eine pandas-UDF namens calculate_shap und übergeben diese Funktion dann an mapInPandas. Diese Methode wird dann verwendet, um die parallelisierte Methode auf den PySpark-Datenrahmen anzuwenden. Wir werden diese UDF verwenden, um unsere SHAP-Performancetests auszuführen.
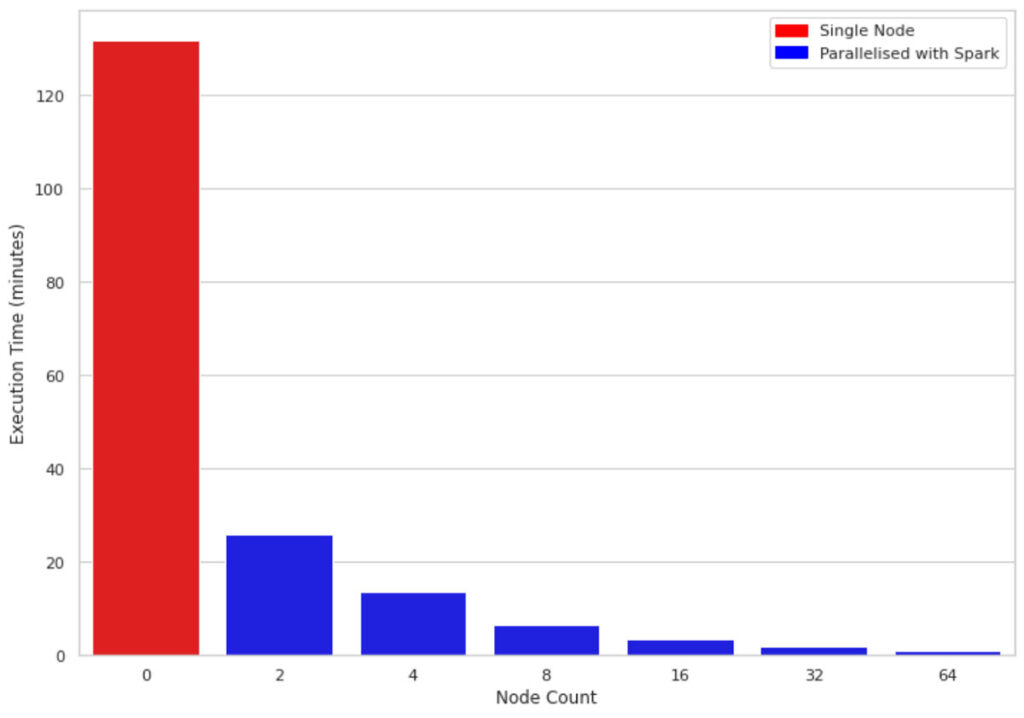
Abbildung 2 vergleicht die Ausführungszeit von 1 Mio. Zeilen und 10 Spalten auf einem Ein-Knoten-Rechner mit clusters der Größen 2, 4, 8, 16, 32 und 64. Die zugrunde liegenden Maschinen sind bei allen Clustern ähnlich (4 Kerne und 30,5 GB Speicher). Eine interessante Beobachtung ist, dass der parallelisierte Code alle Kerne der Knoten im clusters ausnutzt. Daher verbessert selbst ein Cluster der Größe 2 die Performance fast um das Fünffache.
Skalierung bei wachsender Datengröße
Aufgrund der Implementierung von SHAP haben zusätzliche Features einen größeren Einfluss auf die Performance als zusätzliche Zeilen. Jetzt wissen wir, dass SHAP-Werte mit Spark und Pandas UDF schneller berechnet werden können. Als Nächstes sehen wir uns an, wie SHAP mit zusätzlichen Features/Spalten abschneidet.
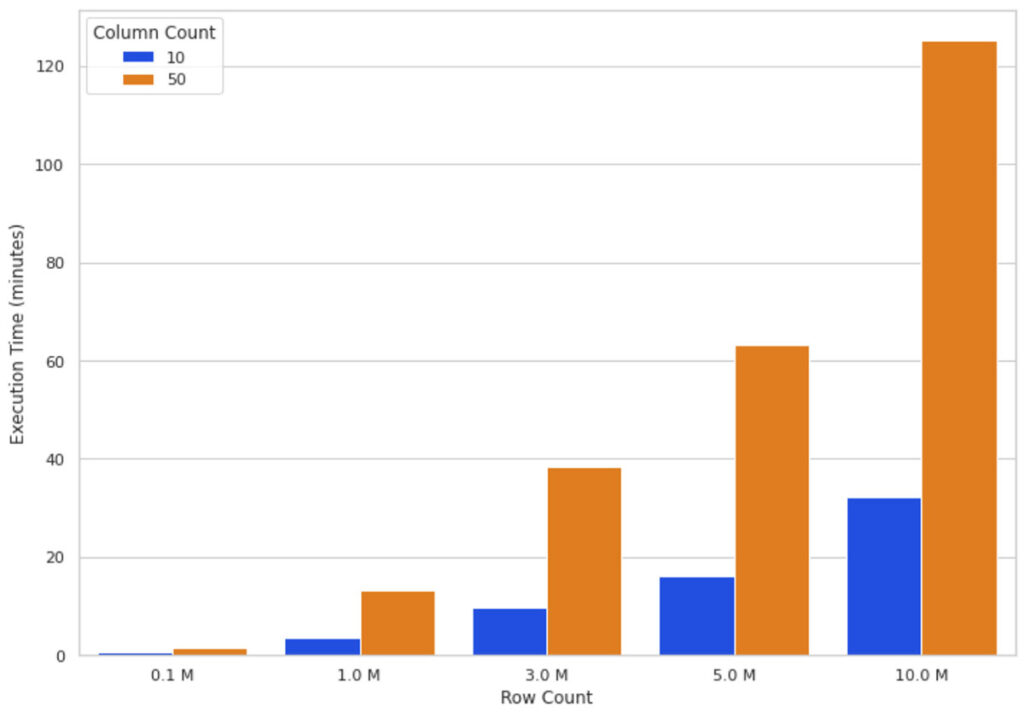
Intuitiv bedeutet eine wachsende Datenmenge, dass der SHAP-Algorithmus mehr Berechnungen durchführen muss. Abbildung 3 zeigt die Ausführungszeiten der SHAP-Werte auf einem 16-Knoten-Cluster für eine unterschiedliche Anzahl von Zeilen und Spalten. Du kannst sehen, dass die Skalierung der Zeilen die Ausführungszeit fast direkt proportional erhöht, d.h. eine Verdopplung der Zeilenzahl verdoppelt die Ausführungszeit fast. Die Skalierung der Anzahl der Spalten steht in einem proportionalen Verhältnis zur Ausführungszeit; das Hinzufügen einer Spalte erhöht die Ausführungszeit um fast 80%.
Diese Beobachtungen (Abbildung 2 und Abbildung 3) führen zu dem Schluss, dass je mehr Daten vorhanden sind, desto besser lässt sich die Berechnung horizontal skalieren (durch Hinzufügen weiterer Worker-Knoten), um die Ausführungszeit in einem angemessenen Rahmen zu halten.
Wann sollte man eine Parallelisierung in Betracht ziehen?
Fragen, die wir beantworten wollten, sind: Wann lohnt sich eine Parallelisierung? Wann sollte man anfangen, PySpark zur Parallelisierung von SHAP-Berechnungen zu verwenden – selbst mit dem Wissen, dass dies den Rechenaufwand erhöhen könnte? Wir haben ein Experiment aufgesetzt, um den Effekt einer Verdopplung der Clustergröße auf die Ausführungszeit der SHAP-Berechnung zu messen. Ziel des Experiments ist es, herauszufinden, ab welcher Datengröße es sich lohnt, mehr horizontale Ressourcen (d. h. weitere Worker-Knoten) für das Problem einzusetzen.
Wir haben die SHAP-Berechnungen für Daten mit 10 Spalten und für Zeilenzahlen von 10, 100, 1000 und so weiter bis zu 10 Mio. ausgeführt. Für jede Zeilenanzahl haben wir die Ausführungszeit der SHAP-Berechnung viermal für Clustergrößen von 2, 4, 32 und 64 gemessen. Das Ausführungszeitverhältnis ist das Verhältnis der Ausführungszeit der SHAP-Werte-Berechnung auf den größeren Clustern (4 und 64) zur Ausführung derselben Berechnung auf einem Cluster mit der halben Anzahl von Knoten (2 bzw. 32).
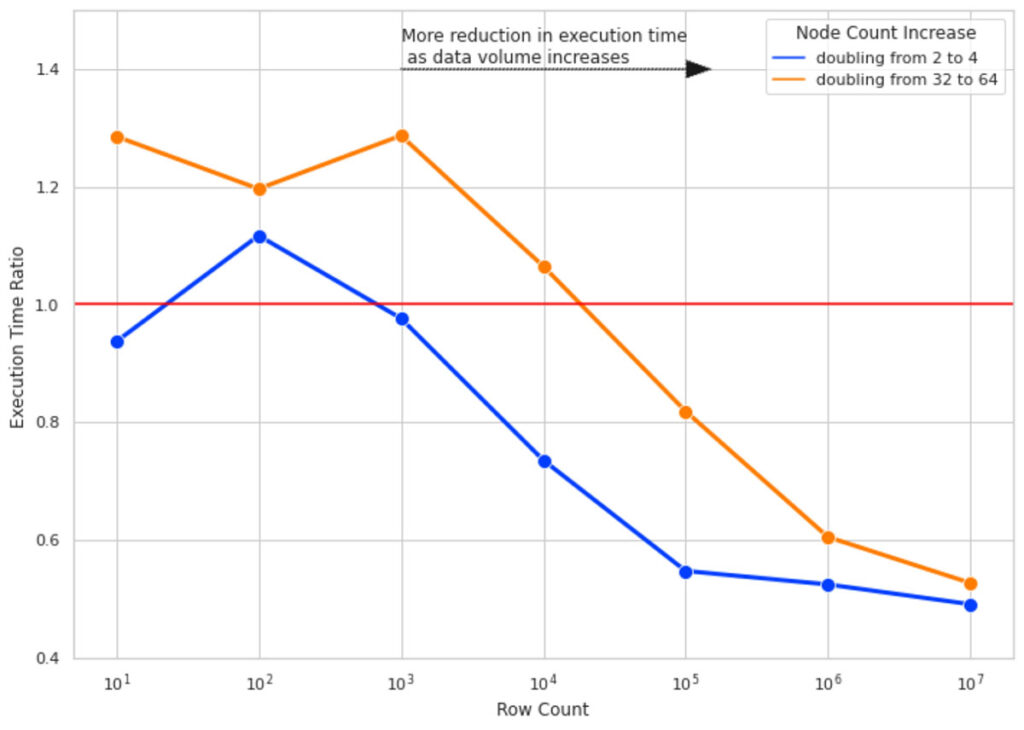
Abbildung 4 veranschaulicht das Ergebnis dieses Experiments. Hier sind die wichtigsten Erkenntnisse:
-
- Bei einer geringen Zeilenanzahl führt die Verdopplung der Clustergröße nicht zu einer Verbesserung der Ausführungszeit, sondern verschlechtert sie in manchen Fällen sogar. Dies ist auf den zusätzlichen Overhead durch das Task-Management von Spark zurückzuführen (daher Ausführungszeitverhältnis > 1).
- Mit zunehmender Zeilenanzahl wird die Verdopplung der clustersize effektiver. Bei 10 Mio. Datenzeilen halbiert eine Verdopplung der Clustergröße die Ausführungszeit fast.
- Bei allen Zeilenanzahlen ist die Verdopplung der Clustergröße von 2 auf 4 effektiver als die Verdopplung von 32 auf 64 (beachten Sie den Abstand zwischen den blauen und orangen Linien). Mit zunehmender Clustergröße nimmt auch der Overhead für das Hinzufügen weiterer Knoten zu. Dies liegt daran, dass bei zu kleinen Datenmengen pro Partition der Overhead für die Erstellung einer separaten Task zur Verarbeitung der geringen Datenmenge größer ist als bei der Verwendung einer optimaleren Daten-/Partitionsgröße.
Gotchas
Repartitioning
Wie oben erwähnt, implementiert Spark Parallelität durch das Konzept der Partitionen; Daten werden in Blöcke von Zeilen partitioniert und jede Partition wird by default von einem einzigen Kern verarbeitet. Wenn Daten anfänglich von Apache Spark gelesen werden, werden möglicherweise nicht unbedingt Partitionen erstellt, die für die Berechnung, die Sie auf Ihrem Cluster ausführen möchten, optimal sind. Insbesondere bei der Berechnung von SHAP-Werten können wir potenziell eine bessere Performance erzielen, indem wir unser Dataset neu partitionieren.
Es ist wichtig, ein Gleichgewicht zu finden, bei dem die Partitionen klein genug sind, aber nicht so klein, dass der Overhead der Partitionen die Vorteile der Parallelisierung der Berechnungen überwiegt.
Für unseren Performancetest haben wir uns entschieden, alle Kerne des Clusters zu nutzen, indem wir den folgenden Code verwendet haben:
Bei noch größeren Datenmengen solltest du die Anzahl der Partitionen auf das 2- oder 3-fache der Anzahl der Kerne einstellen. Der Schlüssel ist, damit zu experimentieren und die beste Partitionierungsstrategie für deine Daten herauszufinden.
Verwendung von display()
Wenn Sie mit einem Databricks Notebook arbeiten, sollten Sie die Verwendung der Funktion display() beim Benchmarking der Ausführungszeiten vermeiden. Die Verwendung von display() zeigt Ihnen nicht unbedingt, wie lange eine vollständige Transformation dauert; sie hat ein implizites Zeilenlimit, das in die Abfrage eingefügt wird, und je nach dem Betrieb, den Sie messen möchten, z. B. das Schreiben in eine Datei, entsteht zusätzlicher Overhead beim Sammeln der Ergebnisse zurück zum Treiber. Unsere Ausführungszeiten wurden mit der write-Methode von Spark unter Verwendung des „noop“-Formats gemessen.
Fazit
In diesem Blogpost haben wir eine Lösung vorgestellt, mit der sich SHAP-Berechnungen durch Parallelisierung mit PySpark und Pandas UDFs beschleunigen lassen. Anschließend haben wir die Performance der Lösung bei zunehmenden Datenmengen, verschiedenen Maschinentypen und wechselnden Konfigurationen bewertet. Hier sind die wichtigsten Erkenntnisse:
-
-
- Die SHAP-Berechnung auf einem einzelnen Knoten wächst linear mit der Anzahl der Zeilen und Spalten.
- Die Parallelisierung von SHAP-Berechnungen mit PySpark verbessert die Performance, indem die Berechnungen auf allen CPUs deines Clusters ausgeführt werden.
- Eine Vergrößerung des Clusters ist effektiver, wenn du größere Datenmengen hast. Bei kleinen Daten ist diese Methode nicht effektiv.
-
Ausblick
Vertikale Skalierung - Der Blogpost sollte zeigen, wie die horizontale Skalierung mit großen Datensätzen die Performance bei der Berechnung der SHAP-Werte verbessern kann. Wir sind davon ausgegangen, dass jeder Knoten in unserem Cluster über 4 Kerne und 30,5 GB verfügt. In Zukunft wäre es interessant, die Performance der vertikalen und horizontalen Skalierung zu testen, z. B. den Vergleich zwischen einem clusters mit 4 Knoten (4 Kerne, je 30,5 GB) und einem clusters mit 2 Knoten (8 Kerne, je 61 GB).
Serialisieren/Deserialisieren – Wie bereits erwähnt, ist einer der Hauptgründe für die Verwendung von Pandas UDFs anstelle von Python UDFs, dass Pandas UDFs Apache Arrow verwendet, um die Serialisierung/Deserialisierung von Daten zwischen dem JVM- und dem Python-Prozess zu verbessern. Es könnte einige potenzielle Optimierungen bei der Konvertierung von Spark-Datenpartitionen in Arrow-Record-Batches geben, wobei das Experimentieren mit der Arrow-Batch-Größe zu weiteren Performance-Steigerungen führen könnte.
Vergleich mit verteilten SHAP-Implementierungen – Es wäre interessant, die Ergebnisse unserer Lösung mit denen von verteilten SHAP-Implementierungen wie Shparkley zu vergleichen. Bei der Durchführung einer solchen Vergleichsstudie wäre es wichtig, zunächst sicherzustellen, dass die Ergebnisse beider Lösungen überhaupt vergleichbar sind.
(Dieser Blogbeitrag wurde mit KI-gestützten Tools übersetzt.) Originalbeitrag
Verpassen Sie keinen Beitrag von Databricks
Was kommt als Nächstes?

Data Science e ML
October 31, 2023/9 min de leitura
